|
Предсказание доли иона пептида заданного заряда в масс-спектрометрии с положительной электроспрейной ионизацией
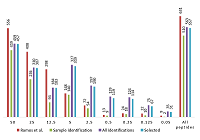
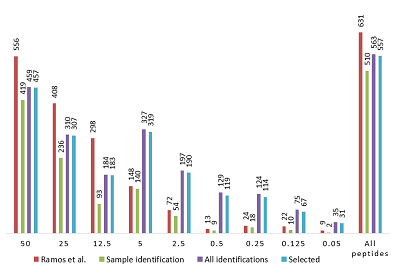
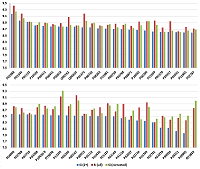
Научно-исследовательский институт биомедицинской химии имени В.Н. Ореховича Ключевые слова: пептид; масс-спектрометрия; электроспрей; предсказание свойств DOI: 10.18097/BMCRM00100 ВВЕДЕНИЕ На настоящий момент масс-спектрометрия (MS) является основным инструментом протеомных исследований [1]. Среди различных вариантов ионизации широко используется такой метод мягкой ионизации, как ионизация электроспреем (ESI). При анализе пептидов, например при тандемной масс-спектрометрии (MS/MS), количество ионов определённого заряда зависит от используемого оборудования, условий эксперимента (приложенное напряжение, концентрация и скорость потока раствора и т.д.), состава растворителя [2]. В то же время очевидно, что распределение ионов при ESI в одних и тех же условиях определяется в первую очередь аминокислотной последовательностью пептида. Таким образом, при планировании эксперимента (например, при выборе способа гидролиза или выборе рабочего окна для регистрации ионов с заданными величинами m/z, при выборе условий отслеживания ионов с определённым зарядом) важно знать, какие зарядные состояния и в какой доле от общего количества пептида можно будет зарегистрировать. Знать долю от общего количества пептида важно также в экспериментах по количественному определению белка с помощью масс-спектрометрических исследований. Конечно, это возможно только при целом ряде допущений: количество различных пептидов при гидролизе примерно совпадет и пропорционально общему количеству белка; «невидимая часть» или нерегистрируемая доля пептидов примерно одинаковая и т.д. Однако представляется, что даже приблизительное предсказание с точностью до 10-15% может качественно улучшить данные масс-спектрометрического эксперимента. В данной работе исследуется возможность предсказания доли иона с конкретным зарядом на основе аминокислотного спектра пептида (последовательности пептида). МЕТОДИКА Наиболее важным фактором в подобного рода работе является наличие качественных, а ещё лучше, и стандартизированных данных. В работе использованы данные, полученные в работе [3], депонированные в ProteomeXchange [4] (идентификатор PXD001819). Данные получены в ходе масс-спектрометрического исследования с использованием стандартизованного набора из 48 высокоочищенных белков человека без SAP (PTM возможны), как синтезированных рекомбинантно, так и полученных из естественных источников и представленных в пробе в одинаковой концентрации (UPS1 компании “Sigma-Aldrich”, США). К особенностям масс-спектрометрического эксперимента относится следующее [3]: 1. пробы для идентификации авторы готовили, смешивая лизат дрожжевых клеток с набором разведений UPS таким образом, чтобы получить конечную концентрацию UPS1 0.05 fmol/mg, 0.125 fmol/mg, 0.250 fmol/mg, 0.5 fmol/mg, 2.5 fmol/mg, 5 fmol/mg, 12.5 fmol/mg, 25 fmol/mg и 50 fmol/mg дрожжевого лизата; 2. гидролиз проводили в растворе c добавлением 2% трипсина; 3. nanoLC-MS/MS проводили с использованием системы nanoRS UHPLC (“Dionex”, Нидерланды, совмещенной с масс-спектрометром LTQ-Orbitrap Velos (“Thermo Fisher Scientific”, Германия) по 3 повтора для каждого разведения; 4. идентификацию пептидов авторы проводили двумя способами: с использованием Mascot Daemon версии 2.4 (“Matrix Science”, Великобритания) с точностью 5 ppm для первичного иона и 0.8 Da для идентификации фрагментов; с использованием комбинации программ MaxQuant [5] и поисковой машины Andromeda [6] (6 ppm и 0.5 Da). В ходе анализа мы совместили данные по идентификации пептидов с данными по распределению первичных ионов различной зарядности (включая суммарную интенсивность всех пиков для данного иона как меру количества детектированных ионов). Данные по всем зарегистрированным первичным ионам были экстрагированы нами из raw-файлов с помощью программы Dinosaur [7], которая кроме суммы интенсивностей всех пиков данного иона вычисляет также начальное время появления и исчезновения данного иона на выходе с хромотографической колонки и время, соответствующее максимальной интенсивности при детекции (время удержания или RT). Затем данные группировали следующим образом. Группы первичных ионов с различным зарядом считали принадлежащим одному пептиду, если масса полного пептида находилась в пределах 5 ppm, времена выхода с колонки перекрывались, а положение максимума отличалось не более чем на 0.2 мин. При сравнении с пептидами, идентифицированными авторами в работе [3], считали, что первичный ион соответствовал идентифицированному пептиду, если значения m/z одинаково заряженных ионов совпадали в пределах 5 ppm, и RT иона, измеренного авторами работы [3], лежало в пределах диапазона RT, вычисленного программой Dinosaur. При формировании выборки распределения первичных ионов по заряду данные усредняли по всем приведенным повторам. При поиске соответствия между данными о распределении первичных ионов и данными по идентификации MS/MS, последние использовались без привязки к конкретному разведению (см. выше п.1). При наличии модификаций данные с N-концевым ацетилированием в выборку не включали, при наличии остатка окисленного метионина использовали только вариант с максимальной суммарной интенсивностью. Данные, полученные в результате такого анализа, представлены в сводной таблице дополнительных материалов. РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ На рисунке 1 представлено распределение числа идентификаций пептидов в каждом из разведений, полученных в работе [3], и при объединении данных в сводную таблицу (дополнительные материалы) по правилам, описанным в разделе «Методика». Видно, что при использовании пептидов, идентифицированных только для соответствующего разведения, существенная часть данных теряется. В общей сложности не найдено соответствия для 121 пептида из 631, идентифицированных в работе [3] (в пределах отдельных разведений различия ещё больше). Из них 69 были обнаружены, но не удовлетворяли установленным параметрам соответствия по точности (5ppm). Ещё 15 надо было исключить из анализа как содержащих запрещённую модификацию (N-концевое ацетилированиe). Учитывая, что во всех опытах условия экспериментов отличались только концентрацией белков UPS1, мы полагаем, что использовать данные о заряде, m/z и RT иона, идентифицированного для другого разведения, правомерно для всех, так как “выбор иона” для фрагментации и получения качественного для идентификации спектра, строго говоря, не детерминирован и может носить случайный характер.
Поскольку суммарная интенсивность - величина, зависимая от настроек прибора и случайных факторов, меняющихся от опыта к опыту, то в качестве меры количества иона с заданным зарядом использовали 2 набора величин. В первом рассматривали долю варианта иона пептида конкретной зарядности (ωn+), вычисленную как отношение суммы интенсивностей данного иона к сумме всех сумм интенсивностей обнаруженных ионов этого пептида. Для любого пептида сумма всех величин ωn+ равна 1. В данной работе рассматривали зарядность ионов от 1+ до 5+ (рис. 1). Были зафиксированы единичные случаи наличия ионов 6+, но они не рассматривались. К сожалению, определить достоверно долю пептидов, не имеющих положительного заряда из эксперимента нельзя. Второй набор величин характеризует соотношение между суммами интенсивностей ионов (Сn/m). В данной работе рассмотрены только 3 пары ионов 1+/2+, 2+/3+ и 3+/4+. Величина Сn/m имеет смысл только в случае существования обоих ионов. Проблема существует и для величины ωn+, если она принимает значение 0 или 1. Нет уверенности, что пептид в подавляющем количестве существует в виде данного иона, или иона не существует вовсе. Возможно этот ион или другие ионы пептида просто не были зарегистрированы, либо неправильно идентифицированы программным обеспечением.
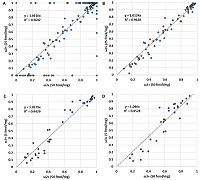
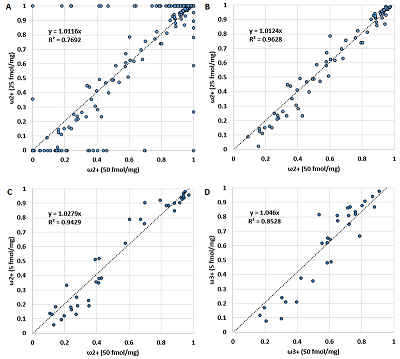
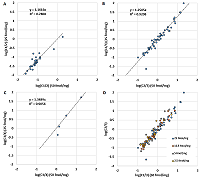
Так как распределение ионов по заряду для отдельного пептида определяется его химической природой (или аминокислотной последовательностью), то оно не должно зависеть от концентрации пептида. Рисунок 2 демонстрирует подтверждение этого постулата. Как и предполагалось изначально, можно наблюдать критические отклонения в случаях, когда величина ωn+ равна 0 или 1. Если отбросить эти значения, то корреляция становится более чем убедительная (табл. 1). Значения Сn/m также не меняются от разведения к разведению (рис. 3, табл. 1). В последнем случае значения для пептидов, соответствующие ωn+, равному 0 или 1, отсутствовали по определению. Таким образом, данные для величин ωn+ и Сn/m можно объединить в одну выборку. Так как наиболее полная выборка - для разведения 50 fmol/mg, то её использовали как базовую, добавляя значения в случае, если пептид обнаруживался только при большем разведении, или заменяя значения в случае, если при большем разведении обнаруживали больше вариантов иона пептида. Если изначально в выборке для разведения 50 fmol/mg было 258 наблюдений с величинами ω2+, отличными от 0 или 1, то в обобщённой выборке таких наблюдения стало 300.
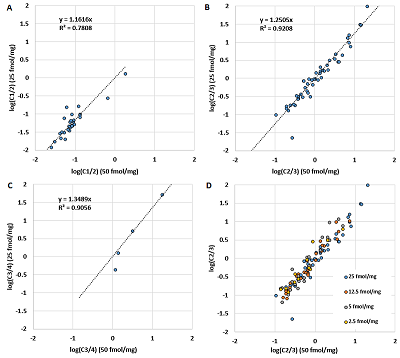
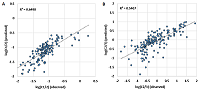
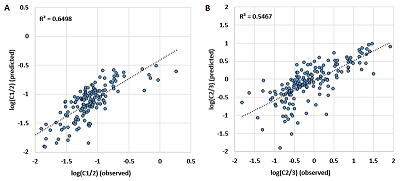
Для создания набора уравнений, с помощью которых можно предсказать распределение ионов по заряду в качестве независимых переменных, использовали аминокислотный спектр пептида (количество каждого из 20 аминокислотных остатков, присутствующих в пептиде). В работе рассматриваются уравнения линейной регрессии. Для данного случая это не оптимальный выбор, так как при использовании линейной регрессии нельзя учесть тот факт, что количественные доли связаны между собой и дают в сумме единицу, а также, что в общем случае не вводятся граничные условия о том, что предсказываемая величина может принимать значения только от 0 до 1. Однако для демонстрации принципиальной возможности такого предсказания линейные уравнения использовать можно. Результаты представлены на рисунке 4 и в таблице 1. Видно, что линейная регрессия даёт удовлетворительный результат во всех случаях, когда количество наблюдений достаточно. Наилучший результат показан для величины Сn/m, особенно в случае С1/2. Больший разброс для величины С2/3, вероятно, связан с тем, что не учитывается наличие ионов с большим зарядом чем 3+, в то время как для С1/2 большая часть наблюдений имеет максимально возможный заряд 2+.
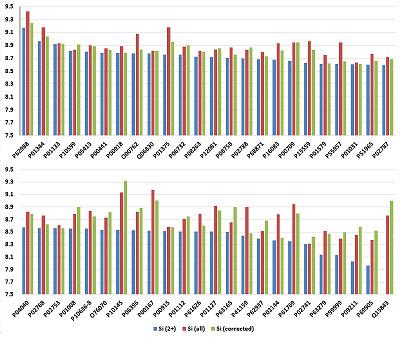
Продемонстрируем пользу такого рода предсказания на примере разведения 50 fmol/mg. На рисунке 5 представлена усреднённая величина Si для каждого из 48 белков набора UPS1. В первом случае были учтены только ионы 2+, наблюдаемые в эксперименте. При этом разброс величины Si лежит в пределах 1.21 логарифмической шкалы, а для одного из белков (48) ионы 2+ не были зарегистрированы. Напомним, что количество каждого белка в наборе UPS1 одинаковое, а усреднение для каждого из них производили, за редким исключением, по четырём и больше пептидам (максимально 28), что должно нивелировать возможные различия, связанные с эффективностью трипсинолиза. Ожидаемым результатом было бы примерное равенство величин Si. Если учитывать суммы Si для всех зарегистрированных ионов, то разброс незначительно уменьшается до 1.12 логарифмической шкалы. Однако если провести коррекцию с использованием предсказания величин С1/2 и С2/3 для наблюдений, в которых были обнаружены только ионы 2+, и величины С2/3, когда обнаруживались исключительно ионы 3+, то разброс уменьшается до 0.9 логарифмической шкалы. При этом эти пептиды заведомо не использовались в обучающей выборке, и, повторимся, линейная регрессия – не лучший метод для решения данной задачи. Тем не менее, даже при использовании очень простых подходов предсказать a priory долю пептида, приходящуюся на ион заданного заряда при ESI, можно.
ФИНАНСИРОВАНИЕ Работа выполнена в рамках государственного задания по программе фундаментальных исследований Государственных академий. ДОПОЛНИТЕЛЬНЫЕ МАТЕРИАЛЫ К данной статье приложены дополнительные материалы, свободно доступные в электронной версии (http://dx.doi.org/10.18097/BMCRM00100) на сайте журнала. ЛИТЕРАТУРА
|