Идентификация генов, мРНК которых подвергаются альтернативному сплайсингу в результате действия эндонуклеазы EndoG в CD4+ т-лимфоцитах человека, мыши и крысы
1Научно-исследовательский институт биомедицинской химии им. В.Н. Ореховича, 119121, Москва, ул. Погодинская, 10; *эл. почта: zhdanovdd@mail.ru 2Российский университет дружбы народов (Университет РУДН), 117198, Москва, ул. Миклухо-Маклая, 6
Ключевые слова: лимфоциты; EndoG; альтернативный сплайсинг; секвенирование
DOI:10.18097/BMCRM00128
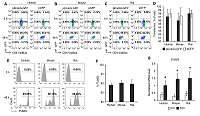
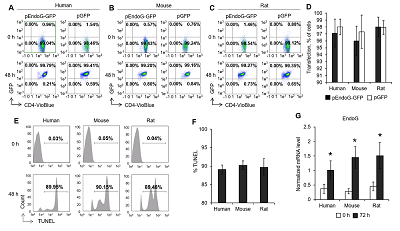
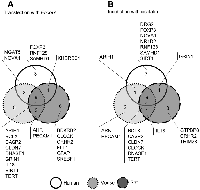
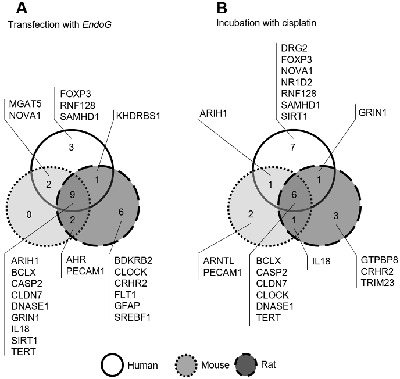
Целью работы явилась идентификация генов, мРНК которых подвергается альтернативному сплайсингу в результате действия апоптотической эндонуклеазы EndoG в нормальных CD4+ Т-лимфоцитах человека, мыши и крысы. С целью индукции EndoG, лимфоциты трансфицировали плазмидой, содержащей ген EndoG, контрольной плазмидой pGFP или инкубировали с цисплатином. Оценивали эффективность трансфекции, количество клеток с повреждением ДНК и уровень мРНК EndoG. Тотальную мРНК клеток секвенировали и оценивали изменение уровней сплайс-вариантов различных генов. Обнаружили изменение пропорции сплайс-вариантов мРНК 28 генов в лимфоцитах человека, мыши и крысы, как в клетках, трансфицированных геном EndoG, так и в клетках, инкубированных с цисплатином. Следовательно, EndoG принимает участие в модуляции альтернативного сплайсинга мРНК идентифицированных генов.
|
ЗАКРЫТЬ

|
Таблица 1.
Идентифицированные гены, АС мРНК которых изменяется при сверхэкспрессии EndoG.
|
ФИНАНСИРОВАНИЕ
Работа выполнена при финансовой поддержке Программы фундаментальных научных исследований государственных академий наук на 2013–2020 годы.
- Kim, E., Magen, A., Ast, G. (2007) Different levels of alternative splicing among eukaryotes. Nucleic Acids Res., 35, 125. DOI
- Zhdanov, D.D., Vasina, D.A., Grachev, V.A., Orlova, E. V., Orlova, V.S., Pokrovskaya, M. V., Alexandrova, S.S., Sokolov, N.N (2017) Alternative splicing of telomerase catalytic subunit hTERT generated by apoptotic endonuclease EndoG induces human CD4 + T cell death. Eur. J. Cell Biol., 96, 653–664. DOI
- Zhdanov, D.D., Vasina, D.A., Orlova, E. V., Orlova, V.S., Pokrovskaya, M. V., Aleksandrova, S.S., Sokolov, N.N. (2017) Apoptotic endonuclease EndoG regulates alternative splicing of human telomerase catalytic subunit hTERT. Biochemistry (Moscow), Suppl. Ser. B Biomed. Chem., 11, 154–165. DOI
- Zhdanov, D.D., Gladilina, Y.A., Pokrovsky, V.S., Grishin, D. V., Grachev, V.A., Orlova, V.S., Pokrovskaya, M. V., Alexandrova, S.S., Plyasova, A.A., Sokolov, N.N. (2019) Endonuclease G modulates the alternative splicing of deoxyribonuclease 1 mRNA in human CD4+ T lymphocytes and prevents the progression of apoptosis. Biochimie, 157, 158–176. DOI
- Zhdanov, D.D., Gladilina, Y.A., Pokrovskaya, M. V., Aleksandrova, S.S., Grishin, D. V., Podobed, O. V., Sokolov, N.N. (2018) Induction of Alternative Splicing and Inhibition of Activity of Telomerase Catalytic Subunit by Apoptotic Endonuclease EndoG in Human T, B, and NK Cells. Bull. Exp. Biol. Med., 164, 478–482. DOI
- Vasina, D.A., Zhdanov, D.D., Orlova, E. V., Orlova, V.S., Pokrovskaya, M. V., Aleksandrova, S.S., Sokolov, N.N. (2017) Apoptotic endonuclease EndoG inhibits telomerase activity and induces malignant transformation of human CD4+ T cells. Biochemistry (Moscow), 82, 24–37. DOI
- Basnakian, A.G., Apostolov, E.O., Yin, X., Napirei, M., Mannherz, H.G., Shah, S. V. (2005) Cisplatin Nephrotoxicity Is Mediated by Deoxyribonuclease I. J. Am. Soc. Nephrol., 16, 697–702. DOI
- Darzynkiewicz, Z., Galkowski, D., Zhao, H. (2008) Analysis of apoptosis by cytometry using TUNEL assay. Methods., 44, 250–254. DOI
- Cheranova, D., Gibson, M., Chaudhary, S., Zhang, L.Q., Heruth, D.P., Grigoryev, D.N., Qing, Ye S. (2013) RNA-seq Analysis of Transcriptomes in Thrombin-treated and Control Human Pulmonary Microvascular Endothelial Cells. J. Vis. Exp., 72, 4393. DOI
- Zhang, L.Q., Cheranova, D., Gibson, M., Ding, S., Heruth, D.P., Fang, D., Ye, S.Q. (2012) RNA-seq Reveals Novel Transcriptome of Genes and Their Isoforms in Human Pulmonary Microvascular Endothelial Cells Treated with Thrombin. PLoS One., 7, e31229. DOI
- Heruth, D.P., Gibson, M., Grigoryev, D.N., Zhang, L.Q., Ye, S.Q. (2012) RNA-seq analysis of synovial fibroblasts brings new insights into rheumatoid arthritis. Cell Biosci., 2, 43. DOI
- Trapnell, C., Roberts, A., Goff, L., Pertea, G., Kim, D., Kelley, D.R., Pimentel, H., Salzberg, S.L., Rinn, J.L., Pachter, L. (2012) Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat. Protoc., 7, 562–578. DOI
- Nebert, D.W. (2017) Aryl hydrocarbon receptor (AHR): "pioneer member" of the basic-helix/loop/helix per-Arnt-sim (bHLH/PAS) family of "sensors" of foreign and endogenous signals. Prog. Lipid Res. 67, 38–57. DOI
- Von Stechow, L., Typas, D., Carreras Puigvert, J., Oort, L., Siddappa, R., Pines, A., Vrieling, H., van de Water, B., Mullenders, L.H.F., Danen, E.H.J. (2015) The E3 Ubiquitin Ligase ARIH1 Protects against Genotoxic Stress by Initiating a 4EHP-Mediated mRNA Translation Arrest, Mol. Mol. Cell. Biol., 35, 1254–1268. DOI
- Buhr, E.D., Takahashi, J.S. (2013) Molecular components of the Mammalian circadian clock. Handb. Exp. Pharmacol., 217, 3–27. DOI
- Bruey, J.-M., Bruey-Sedano, N., Luciano, F., Zhai, D., Balpai, R., Xu, C., Kress, C.L., Bailly-Maitre, B., Li, X., Osterman, A., Matsuzawa, S., Terskikh, A. V., Faustin, B., Reed, J.C. (2007) Bcl-2 and Bcl-XL Regulate Proinflammatory Caspase-1 Activation by Interaction with NALP1. Cell., 129, 45–56. DOI
- Falsetta, M.L., Foster, D.C., Woeller, C.F., Pollock, S.J., Bonham, A.D., Haidaris, C.G., Phipps, R.P. (2016) A Role for Bradykinin Signaling in Chronic Vulvar Pain. J. Pain., 17, 1183–1197. DOI
- Zhivotovsky, B., Orrenius, S. (2005) Caspase-2 function in response to DNA damage. Biochem. Biophys. Res. Commun., 331, 859–867. DOI
- Kazuo, M., Sumio, S. (1994) Oligo-capping: a simple method to replace the cap structure of eukaryotic mRNAs with oligoribonucleotides. Gene, 138, 171–174. DOI
- Hardin, P.E. (2000) From biological clock to biological rhythms. Genome Biol., 1 (4), reviews1023.1 - reviews1023.5. DOI
- Bale, T.L., Vale, W.W. (2004) CRF AND CRF RECEPTORS: Role in Stress Responsivity and Other Behaviors. Annu. Rev. Pharmacol. Toxicol., 44, 525–557. DOI
- Peitsch, M.C., Polzar, B., Stephan, H., Crompton, T., MacDonald, H.R., Mannherz, H.G., Tschopp, J. (1993) Characterization of the endogenous deoxyribonuclease involved in nuclear DNA degradation during apoptosis (programmed cell death). EMBO J., 12, 371–377. DOI
- Ko, M.S., Lee, U.H., Kim, S. I., Kim, H.J., Park, J.J., Cha, S.J., Kim, S.B., Song, H., Chung, D.K., Han, I.S., Kwack, K., Park, J.W. (2004) Overexpression of DRG2 suppresses the growth of Jurkat T cells but does not induce apoptosis. Arch. Arch. Biochem. Biophys., 422, 137–144. DOI
- Lu, L., Barbi, J., Pan, F. (2017) The regulation of immune tolerance by FOXP3. Nat. Rev. Immunol., 17, 703–717. DOI
- Ji, S., Xin, H., Li, Y., Su, E.J. (2018) FMS-like tyrosine kinase 1 (FLT1) is a key regulator of fetoplacental endothelial cell migration and angiogenesis. Placenta., 70, 7–14. DOI
- Middeldorp, J., Hol, E.M. (2011) GFAP in health and disease. Prog. Neurobiol., 93, 421–443. DOI
- Scala, M., Amadori, E., Fusco, L., Marchese, F., Capra, V., Minetti, C., Vari, M.S., Striano, P. (2019) Abnormal circadian rhythm in patients with GRIN1-related developmental epileptic encephalopathy. Eur. J. Paediatr. Neurol., 23, 657–661. DOI
- Vizeacoumar, F.J., Arnold, R., Vizeacoumar, F.S., et al. (2013) A negative genetic interaction map in isogenic cancer cell lines reveals cancer cell vulnerabilities1. Mol. Syst. Biol., 9, 696. doi:10.1038/msb.2013.54 DOI
- Yasuda, K., Nakanishi, K., Tsutsui, H. (2019) Interleukin-18 in Health and Disease. Int. J. Mol. Sci., 20, E649. DOI
- Fu, K., Sun, X., Wier, E.M., Hodgson, A., Liu, Y., Sears, C.L., Wan, F. (2016) Sam68/KHDRBS1 is critical for colon tumorigenesis by regulating genotoxic stress-induced NF-κB activation. Elife, 5, e15018. DOI
- Deng, Q., Chen, Y., Yin, N., Shan, N., Luo, X., Yuan, Y., Luo, X., Liu, Y., Liu, X., Qi, H. (2017) The Role of MGAT5 in Human Umbilical Vein Endothelial Cells. Reprod. Sci., 24, 313–323. DOI
- Kim, E.K., Cho, Y.A., Seo, M.-K., Ryu, H., Cho, B.C., Koh, Y.W., Yoon, S.O. (2019) NOVA1 induction by inflammation and NOVA1 suppression by epigenetic regulation in head and neck squamous cell carcinoma. Sci. Rep., 9, 11231. DOI:10.1038/s41598-019-47755-8 DOI
- Yin, L., Wu, N., Curtin, J.C., Qatanani, M., Szwergold, N.R., Reid, R.A., Waitt, G.M., Parks, D.J., Pearce, K.H., Wisely, G.B., Lazar, M.A. (2007) Rev-erb, a Heme Sensor That Coordinates Metabolic and Circadian Pathways. Science, 318, 1786–1789. DOI
- Lertkiatmongkol, P., Liao, D., Mei, H., Hu, Y., Newman, P.J. (2016) Endothelial functions of platelet/endothelial cell adhesion molecule-1 (CD31). Curr. Opin. Hematol, 23, 253–259. DOI
- Anandasabapathy, N., Ford, G.S., Bloom, D., Holness, C., Paragas, V., Seroogy, C., Skrenta, H., Hollenhorst, M., Fathman, C.G., Soares, L. (2003) GRAIL: an E3 ubiquitin ligase that inhibits cytokine gene transcription is expressed in anergic CD4+ T cells. Immunity., 18, 535–547. DOI
- Berger, A., Sommer, A.F.R., Zwarg, J., Hamdorf, M., Welzel, K., Esly, N., Panitz, S., Reuter, A., Ramos, I., Jatiani, A., Mulder, L.C.F., Fernandez-Sesma, A., Rutsch, F., Simon, V., König, R., Flory, E. (2011) SAMHD1-deficient CD14+ cells from individuals with Aicardi-Goutières syndrome are highly susceptible to HIV-1 infection. PLoS Pathog., 7, e1002425. DOI
- Sun, C., Zhang, F., Ge, X., Yan, T., Chen, X., Shi, X., Zhai, Q. (2007) SIRT1 improves insulin sensitivity under insulin-resistant conditions by repressing PTP1B. Cell Metab., 6, 307–319. DOI
- Ferré, P., Foufelle, F. (2010) Hepatic steatosis: a role for de novo lipogenesis and the transcription factor SREBP-1c. Diabetes, Obes. Metab., 12, 83–92. DOI
- Liu, X., Wang, Y., Chang, G., Wang, F., Wang, F., Geng, X. (2017) Alternative Splicing of hTERT Pre-mRNA: A Potential Strategy for the Regulation of Telomerase Activity. Int. J. Mol. Sci., 18(3), 567 DOI
- Sparrer, K.M.J., Gableske, S., Zurenski, M.A., Parker, Z.M., Full, F., Baumgart, G.J., Kato, J., Pacheco-Rodriguez, G., Liang, C., Pornillos, O., Moss, J., Vaughan, M., Gack, M.U. (2017) TRIM23 mediates virus-induced autophagy via activation of TBK1. Nat. Microbiol., 2, 1543–1557. DOI