|
Обобщённая модель предсказания константы ингибирования мускариновых холинорецепторов M1-M5
1Научно-исследовательский институт биомедицинской химии имени В. Н. Ореховича, Ключевые слова: мускариновые ацетилхолиновые рецепторы; ингибиторы; конкурентное ингибирование; докинг; вычислительные методы; молекулярная динамика; QSAR DOI: 10.18097/BMCRM00129 ВВЕДЕНИЕ
Ацетилхолиновые мускариновые рецепторы (М-холинорецепторы) представляют собой группу белков, которые принимают участие в регуляции когнитивных процессов, оказывают влияние на работу желез внутренней секреции, управляют работой гладкой мускулатуры, а также контролируют двигательную активность [1]. Разные варианты (подтипы) рецепторов имеют различную локализацию в тканях организма и часто рассматриваются как важные биомишени, ингибиторы которых можно использовать в качестве лекарственных средств для лечения целого спектра патологических состояний [2]. Причём, важно рассматривать селективность связывания того или иного соединений (потенциального или реального лекарственного средства), чтобы избежать возможных побочных реакций в организме [3]. Ранее нами уже была опубликована работа, в которой исследована возможность создания набора компьютерных моделей для предсказания набора констант ингибирования (Ki) для четырёх из пяти М-холинорецепторов (M1-M4) [4]. В работе использовали моделирование комплексов методом молекулярного докинга с последующей симуляцией молекулярной динамики и создание линейных уравнений, основанных на энергетических параметрах, рассчитанных методом MM-PBSA/MM-GBSA. Однако за прошедшее время в Protein Data Bank (PDB) [5] стала доступна 3D структура M5 рецептора [6]. Кроме того, в настоящей работе использована расширенная выборка соединений – ингибиторов M1-M5 рецепторов, а также введены дополнительные правила отбора вариантов докирования, основанные на априорном знании о структуре комплексов с гомологичными лигандами. Основной задачей, как и в предыдущей работе, было создание набора моделей для оценки параметров конкурентного ингибирования относительно метил-скополамина произвольного лиганда к набору М-холинорецепторов (M1, M2, M3, M4 и M5). МАТЕРИАЛЫ И МЕТОДЫ 3D cтруктуры рецепторов и подготовка данных На момент подготовки статьи из 5 М-холинорецепторов человека 4 (M1, M2, M4 и M5) имели известную 3D-структуру, доступную в PDB [6-8] (табл. 1).
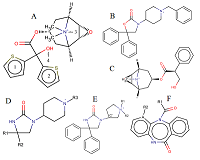
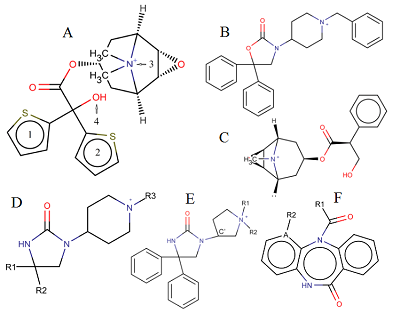
Кроме того, известна структура ещё одного рецептора (M3-холинорецептора) крысы [9]. Как видно из таблицы 1, аминокислотные последовательности рецепторов существенно различаются. Следует также отметить, что трёхмерная структура, строго говоря, получена для химерных рецепторов, у которых внемембранная часть заменена на фрагмент другого белка, который, в свою очередь, позволяет сохранить архитектуру мембранной части рецептора [6-9]. Только структуры рецепторов M1 (код в PDB 5CXV) и M2 (3UON) пригодны для молекулярного моделирования без дополнительных подготовительных процедур. Для рецепторов M4 (5DSG) и M5 (6OL9) элементы петель с неразрешёнными координатами были заделаны простым подбором петель, подходящих по конфигурации, средствами программы SYBYL-X [10]. Аминокислотные остатки, входящие в состав данных петель, находятся далеко от места связывания, и их реконструкция обеспечивает восстановление целостности структуры, что является необходимым условием для проведения молекулярной динамики, которая играет большую роль в нашей работе. Структура M3 холинорецептора человека была построена ранее на основании рецептора крысы (4DAJ) [4]. Важно, что 4 из 5 рецепторов представлены в виде комплексов с тиотропием (Tiotropium, (1α,2β,4β,7β)-7-[(гидроксиди-2-тиэнилацетил)окси]-9,9-диметил-3-окса-9-азониатрицикло[3.3.1.02,4]нонан), а рецептор M2 с соединением (3R)-1-азабицикло[2.2.2]окт-3-ил(гидрокси)ди(фенил)ацетат ((3R)-1-azabicyclo[2.2.2]oct-3-yl hydroxy(diphenyl)acetate), хоть и отличным химически, но имеющим общие (или схожие по свойствам) с тиотропием структуры, позволяющие выделить набор фармакофорных мотивов, положение которых в комплексе может быть использовано как вторичный фильтр для отбора гипотез положения лиганда, полученных молекулярным докингом. В нашей работе использовали (рис.1А): 2 преимущественно гидрофобных цикла, положительно заряженный атом азота и положение OH группы в центральной части молекулы. Выбор обусловлен тем, что у большинства структур, использованных в работе, можно было выделить как минимум три аналогичных фармакоформных фрагмента (рис. 1). В основном были представлены все четыре, но в отдельных случаях отсутствовал один из циклов, либо центральная OH-группа при наличии карбонильной (в качестве фармакофорной точки использовали положение атома кислорода). Поскольку все имеющиеся структуры представляют из себя комплексы с известными ингибиторами, то подготовка включала в себя простую минимизацию в поле сил TRIPOS [10] для расчёта электростатических взаимодействий для рецептора; в дальнейшем для низкомолекулярных ингибиторов использовалась схема частичных зарядов из мерковского поля сил [10].
Данные по ингибиторам с известными значениями Ki. В настоящей работе были использованы 3 набора данных для нескольких групп соединений [11-13] (см. дополнительные материалы), отличительной особенностью которых является то факт, что для большей части значения Ki известны для 2-х холинорецепторов (M2 и M3), а для примерно трети из них - для трёх-пяти холиновыхрецепторов человека. Примеры базовых структур представлены на рисунке 1. Важно, все данные были получены на одном наборе клеточных линий, и величина Ki была оценена при помощи одного метода [14] – ингибированию связывания меченого метилскополамина (Всего для M1 холинорецептора известно 34 наблюдения, при этом диапазон значений pKi, используемый для дальнейших расчётов, составлял от 6.7 до 9.8 логарифмических единиц. Для выборок M2-M5 известны соответственно 86 наблюдений (диапазон pKi 5.3-12.5), 108 (6.1-12.5), 28 (5.9-10.3) и 28 (5.4-9.9) наблюдений. Общим недостатком выборок для M1, M4 и M5 холинорецепторов были их небольшие размеры. Для всех выборок также имеется проблема с неравномерным распределением данных, большая часть которых соответствует узкому диапазону в 2 логарифмические единицы. Процедура моделирования комплексов и расчёта параметров для создания предсказательных моделей Комплексы ингибиторов с рецепторами моделировали по единой схеме, уже описанной ранее [4], с некоторыми модификациями:
Для построения предсказательных моделей были использованы простые уравнения линейной регрессии. Так как в результате для таких уравнений было использовано 9 независимых переменных, то малые размеры выборок для , не позволяющим остановиться на 5 независимых уравнениях. Выборки сливали между собой, однако при этом число независимых переменных увеличивалось за счёт введения индикаторных переменных, принимавших значение 1, если наблюдение бралось из выборки для конкретного рецептора и 0 для остальных рецепторов (так, если в выборки объединяли данные для 3 рецепторов, добавляли 2 индикаторные переменные). Для выполнения расчетов был использован гибридный высокопроизводительный вычислительный комплекс Федерального исследовательского центра «Информатика и управление» Российской Академии Наук (ФИЦ ИУ РАН) [18] IBM на базе CPU Power9 и графических ускорителей Nvidia Tesla V100. РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ Основные результаты работы на различных этапах представлены в таблице 2. Важно отметить, что в работе не ставилась задача любой ценой получить комплекс лиганда с белком, удовлетворяющий условию сходства по положению в кристалле с тиотропием для определённых фармакофорных мотивов. Более того, например, для соединений, соответствующих базовой структуре на рисунке 1F, сделать это однозначно нельзя (трёхчленный цикл имеет заместители с выраженной асимметрией, дающие как минимум два варианта положения в кристалле), так что они были вынуждено отброшены, чтобы не привносить в модели элемент произвола. При тестировании подобной группы соединений следует рассматривать несколько групп вариантов, содержащих больше фармакофорных элементов. Всего после процедуры докирования из 284 возможных вариантов комплексов было отобрано 199, причём, автоматизированный докинг не нашёл решения для 17 вариантов, а остальные 68 не совпадали с заданным расположением фармакофорных групп.
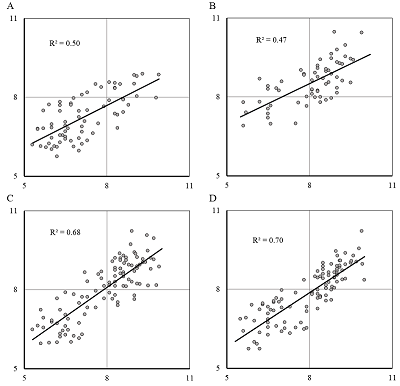
Рассмотрим сначала результаты для отдельных выборок (табл. 2). Результаты для выборок M1, M4 и М5 не могут считаться значимыми, так как, несмотря на хорошие показатели при обучении (кроме M1), тест на случайное перемешивание данных показывает величину усреднённого R2 в диапазоне от 0.3 до 0.51. Косвенно это указывает, что в данном случае возможно переобучение, связанное с малыми размерами и неравномерностью распределения данных. Для выборок M1 и M5 об этом свидетельствует и процедура скользящего контроля методом выбрасывания по одному (Q2 меньше 0.2). Для выборок M2 и M3 этот эффект отсутствует (усреднённый R2 обучения при случайном перемешивании 0.09 и 0.12 соответственно). Процедура скользящего контроля показывает результат ниже порогового значения (Q2=0.6), но близкий к нему. В тоже время тест на перекрёстное предсказание в ряде случаев демонстрирует результат даже лучше ожидаемого (например, предсказание для выборок M2 и M3 по уравнению, построенному для выборки M4). Это позволило ожидать, что при слиянии выборок можно получить результат, как минимум, не хуже. Как видно из таблицы 2, проблема переобучения, связанная с размерами выборки, для комбинированных выборок отсутствовала, несмотря на то, что число независимых переменных было увеличено. Однако, если в общей выборке отсутствовали данные из выборок M2 или M3, сохранялась существенная неравномерность в распределении данных. Так как тестовой выборки для суммирующей данные по всем 5 рецепторам не было, то провели контрольный эксперимент, разделив её пополам на чётные и нечётные номера после ранжирования по значению pKi. Для выборок, в которых полностью отсутствовали данные для одного из рецепторов, R2 предсказания был около 0.5 (рис. 2A и 2B), что уже позволяет отличить сильные ингибиторы холинорецепторов от слабых. Деление же общей выборки пополам (R2 предсказания не меньше 0.68, рис. 2С и 2В) позволяет ожидать, что для схожих по структуре химических соединений, при условии наложения дополнительного ограничения при отборе вариантов после докинга, можно ранжировать их как минимум на 3 класса: сильные, средние и слабые ингибиторы. Так как различные комбинации выборок дают в двух типах тестовых экспериментов R2 предсказания на уровне от 0.6 до 0.7, то это, вероятно, предельная точность предсказания, которую можно получить на рассматриваемом наборе данных. При этом среднее значение ошибки предсказания 0.55 единицы логарифмической шкалы, а максимальная ошибка 1.65 единицы при ширине диапазона значений pKi в 4.7 единицы.
Таким образом, было показано, что внесение априорного знания в процедуру отбора вариантов докирования позволяет в случае ацетилхолиновых мускариновых рецепторов построить общую модель предсказания величины pKi на основе особенностей связывания химического соединения с конкретным рецептором. СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВ Настоящая статья не содержит каких-либо исследований с участием людей или с использованием животных в качестве объектов. ФИНАНСИРОВАНИЕ Работа выполнена в рамках государственного задания на 2020 год (тема № 0090-2017-0020). Адаптация программного обеспечения под гибридную платформу Power9 в ЦОД ФИЦ ИУ РАН выполнена при поддержке гранта РФФИ № 18-29-03100. КОНФЛИКТ ИНТЕРЕСОВ Авторы заявляют об отсутствии конфликта интересов. ДОПОЛНИТЕЛЬНЫЕ МАТЕРИАЛЫ К данной статье приложены дополнительные материалы, свободно доступные на сайте журнала (http://dx.doi.org/10.18097/BMCRM00129). ЛИТЕРАТУРА
|