|
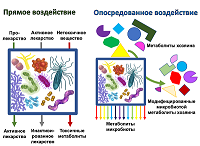
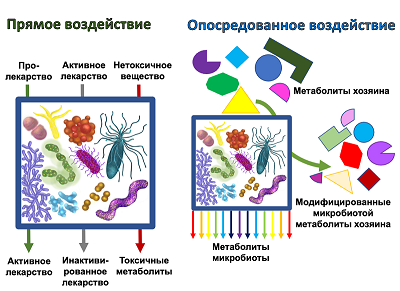
СОДЕРЖАНИЕ 1. ПРЯМАЯ БИОТРАНСФОРМАЦИЯ ЛЕКАРСТВЕННЫХ СОЕДИНЕНИЙ 2. ОПОСРЕДОВАННОЕ ВЛИЯНИЕ МИКРОБИОТЫ НА ЛЕКАРСТВЕННУЮ ТЕРАПИЮ 3. ВОЗМОЖНОСТИ МОДЕЛИРОВАНИЯ МЕТАБОЛИЗМА ЛЕКАРСТВЕННЫХ СОЕДИНЕНИЙ СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВ Рисунок 1Влияние микробиоты на метаболиты хозяина и ксенобиотики Таблица 1Типы химических реакций при метаболизме ксенобиотиков. |
Микробиом кишечника и метаболизм лекарственных соединений
Федеральный научно-клинический центр физико-химической медицины ФМБА, Ключевые слова: микробиота; метагеном; биотрансформация; ксенобиотики; микрофлюидика; вычислительное моделирование DOI: 10.18097/BMCRM00146 ВВЕДЕНИЕ Эффективность фармакотерапии существенно отличается у разных людей. Однако регистрируемых генетических различий между людьми недостаточно для объяснения всего спектра наблюдаемых отличий в ответах на те или иные лекарственные препараты [1]. Поскольку большинство лекарственных препаратов принимается перорально, именно кишечная микробиота, представляющая наиболее разнообразное и многочисленное микробное сообщество, стала объектом пристального внимания исследователей. Возросший интерес к оценке влияния микробиоты на метаболизм лекарственных соединений сформировал новое направления науки – фармакомикробиомику, получившей свое развитие совместно с фармакогеномикой. Данные многочисленных метагеномных исследований свидетельствуют, что количество эукариотических и бактериальных клеток в организме человека сопоставимо – около 3.0∙1013 [2]; при этом количество генов в метагеноме кишечника человека составляет около 530 000 против 30 000 в геноме человека [3]. Согласно теории функциональной избыточности [4], кишечная экосистема образована “бактериальным супервидом” с очень большим геномом, состоящим из широко расходящихся микробных линий, чьи геномы содержат функционально сходные наборы генов, которые могут привести к возникновению согласованного единого метаболического исхода. Рассматривая вопрос влияния на метаболизм лекарственных средств, можно говорить о различных механизмах, в которых прямо или косвенно участвует микробиота кишечника. Прямое воздействие включает в себя биотрансформацию, которое приводит к изменению химической структуры лекарственного соединения, вплоть до деградации вещества [5]. В ряде случаев биотрансформация приводит к преобразованию пролекарства в активный препарат. Однако в других случаях препарат инактивируется или же преобразуется в токсичную форму. Схематично варианты таких взаимодействий представлены на рисунке 1.
1. ПРЯМАЯ БИОТРАНСФОРМАЦИЯ ЛЕКАРСТВЕННЫХ СОЕДИНЕНИЙ Биотрансформация ксенобиотиков является одной из основных функций кишечной микробиоты. Кишечник, как тонкий, так и толстый, обеспечивает среду с низким окислительно-восстановительным потенциалом и отсутствием или малым количеством доступного кислорода. Такие анаэробные и восстановительные условия в кишечнике опосредуют восстановительный метаболизм поступающих ксенобиотиков резидентными кишечными бактериями. Перорально вводимые лекарственные средства могут метаболизироваться кишечной микробиотой (например, путем восстановления) и/или ферментами, метаболизирующими лекарственные средства хозяина (путем окисления), вследствие чего они биоактивируются, элиминируются или инактивируются. С другой стороны, показано, что использование лекарств связано с наибольшей межиндивидуальной изменчивостью микробиоты кишечника, и многие неантибиотические препараты, как установлено, обладают антикомменсальной активностью, предполагая, что некоторые лекарства вызывают изменения состава микробиоты кишечника. С точки зрения роли кишечной микробиоты в поддержании здоровья хозяина или развитии заболеваний, некоторые отпускаемые по рецепту лекарства (и другие ксенобиотики) могут непреднамеренно влиять на физиологию хозяина через их воздействие на состав/структуру кишечной микробиоты. О метаболизме ксенобиотиков кишечной микробиотой сообщалось на протяжении десятилетий и подтверждалось доказательствами, представленных для лекарственных препаратов различных химических классов и терапевтического действия. Сегодня мы можем утверждать, что микробиота кишечника как единая экосистема содержит ферменты, обеспечивающие каскады химических реакций, которые приводят к биотрансформации ряда соединений (табл. 1).
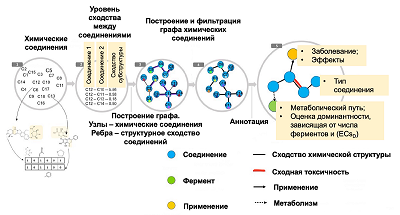
Однако, несмотря на накопленные свидетельства происходящей под действием кишечной микробиоты биотрансформации ксенобиотиков, биохимические и генетические основы этих процессов до сих пор в основном неизвестны. Отчасти это связано с ощутимыми трудностями культивирования некоторых кишечных бактерий и чрезвычайной сложностью сообщества кишечной микробиоты. Для некоторых лекарств были идентифицированы и охарактеризованы ферменты определённых кишечных бактерий, ответственные за их биотрансформацию, но для большинства соединений доступны только косвенные доказательства, указывающие на опосредованный кишечными бактериями восстановительный метаболизм. Физиологические роли даже известных кишечных бактериальных ферментов до конца не определены. При этом вклад микробиоты кишечника в биотрансформацию препарата подчас доминирует над метаболическими процессами, протекающими под действием ферментов хозяина. Zimmermann и соавторы обнаружили, что индивидуальные реакции на препарат зависят от генетики микробиоты человека [7]. Они исследовали метаболизм нуклеозидных препаратов, которые используются в качестве противовирусных и антидепрессантов, у мышей, имеющих различные мутантные микробиоты. Затем они смоделировали фармакокинетику в различных частях тела и определили вклад хозяина и микроорганизмов. Было выявлено, что у некоторых мышей до 70% биотрансформации лекарств может быть отнесено к микробному метаболизму [7]. Другим следствием из взаимосвязи генетических особенностей микробиоты и реакции организма хозяина на лечение является еще один подход к поиску терапии: влияние на экспрессию тех или иных генов у конкретных бактерий. Singh и соавторы было показали [8], что обработка изолятов нескольких бактерий с множественной лекарственной устойчивостью иммуно-антибиотиком двойного-действия форме пролекарства дает эффект, аналогичный условному нокаутированию целевого гена. Также авторы отмечают, что это исследованное на мышиной модели пролекарство (ингибитор 4-гидрокси-3-метилбут-2-енилдифосфатредуктазы, EC 1.17.1.2) стимулировало иммунную реакцию организма хозяина. Исходя из этого можно предположить, что перспективным направлением разработок лекарств является создание комбинации из самого действующего вещества и вспомогательного вещества, модулирующего экспрессию определенных генов конкретных бактерий, продукты которых способствуют снижению эффективности препарата. Как известно, микробиом кишечника человека метаболизирует многие лекарственные средства [6]. Zimmermann с коллегами провели испытания 76 штаммов бактерий из кишечника человека, представляющих 68 видов из основных таксономических групп бактерий, метаболизировать 271 препарат in vitro [9]. Эти препараты были выбраны для обеспечения разнообразности группы с точки зрения таких аспектов, как молекулярная структура или воздействие на организм. Было обнаружено, что 176 из исследованных препаратов претерпели значительные метаболические изменения, вызванные хотя бы одним бактериальным штаммом, что привело к снижению уровня активности молекулы препарата. Каждый протестированный штамм бактерий метаболизировал хотя бы некоторые из препаратов. При этом количество препаратов на штамм варьировало от 11 до 95. Учитывая, что авторы тестировали широко репрезентативную группу лекарственных средств, масштабы этих результатов примечательны, поскольку они повышают вероятность того, что большинство лекарственных средств модифицируются микробиотой. После проведения анализа продуктов биотрансформации 176 препаратов с помощью масс-спектрометрии, было обнаружено 868 уникальных молекул. Эти данные указывают на то, что в результате биотрансформации кишечными бактериями из некоторых лекарственных средств могут быть получены более одного метаболита. Достаточно хорошо изучены пути взаимодействия микробиоты и различных классов гипотензивных препаратов. Антигипертензивные препараты, такие как амлодипин и нифедипин, метаболизируются микробными ферментами кишечника, что может влиять на всасывание лекарств, что приводит к изменению фармакологической активности препарата и возможному отказу от надлежащего контроля артериального давления или неожиданным побочным эффектам. Это, вероятно, указывает на наличие дополнительных механизмов, которые изменяют терапевтическую эффективность гипотензивных препаратов, особенно при определённых патологических состояниях желудочно-кишечного тракта или при одновременном применении таких веществ, как антибиотики и пробиотики, которые могут изменять микробный состав кишечника. Большинство гидрофобных ксенобиотиков, вводимых перорально, превращаются в гидрофильные продукты посредством окисления и конъюгации для облегчения их выведения и детоксикации. Эти процессы происходят в основном в печени. Однако обнаружено, что ксенобиотики, вводимые перорально, также могут метаболизироваться кишечными микробными ферментами, прежде чем всасываться из желудочно-кишечного тракта в кровь. Метаболические реакции, выполняемые микробиотой кишечника, значительно отличаются от метаболических реакций печени. Микробиота желудочно-кишечного тракта в основном метаболизирует лекарственные препараты и фитохимические веществ до гидрофобных соединений, что влияет на всасывание лекарств и может изменять их фармакологические эффекты. В этом участвуют основные микробные ферменты, такие как β-глюкозидазы, β-глюкуронидазы, сульфатазы, азоредуктазы, нитроредуктазы и нитратредуктазы. Типичные примеры соединений и лекарственных препаратов, метаболизируемых микробиотой кишечника, включают симвастатин, пронтозил, иринотекан, глицирризин, амигдалин, байкалеин, гинсенозиды и генистеин. Некоторые препараты, такие как ацетаминофен, амлодипин, хлорамфеникол, дигоксин, ловастатин и сульфасалазин, также подвержены влиянию кишечного микробного метаболизма (табл. 1). Несмотря на то, что метаболизм ксенобиотиков микробными ферментами кишечника до некоторой степени известен, информация о конкретной микрофлоре, ферменты которой катализируют каждую метаболическую реакцию, всё ещё ограничена. Микробиота оказывает сложное влияние на метаболизм и транспорт вводимых перорально лекарственных соединений и на метаболическую активность слизистой оболочки кишечника. Перорально вводимые препараты, особенно те, которые не всасываются полностью из тонкой кишки или кандидаты на энтерогепатическую циркуляцию, неизбежно соприкасаются с микробиотой дистального отдела кишечника. Микробиота кишечника обладает разнообразной и обширной способностью к биотрансформации для метаболизма ксенобиотических соединений. Условия, влияющие на микробный профиль кишечника, такие как стерилизация животных или лечение антибиотиками, могут приводить к изменениям фармакокинетики ксенобиотических соединений, включая фитохимические вещества, диетические соединения и многие лекарственные препараты. Хотя в весьма ограниченном количестве исследований непосредственно изучалось влияние микробиоты кишечника на фармакокинетику гипотензивных препаратов, известно, что микробиота влияет на биодоступность гипотензивных препаратов различными путями, включая метаболизм бактерий, модуляцию ферментов метаболизма и кишечный транспорт. Такое вовлечение микробиоты кишечника может указывать на дополнительные механизмы, которые могут изменять терапевтическую эффективность гипотензивных препаратов, особенно при определённых патологических состояниях желудочно-кишечного тракта или при одновременном применении таких веществ, как антибиотики и пробиотики, которые могут изменять микробный состав кишечника. В свете этих возможностей в будущих исследованиях будут уделять больше внимания потенциальному метаболизму гипотензивных препаратов микробиотой кишечника [10]. Благодаря достижениям в области метагеномики, метаболомики и биоинформатики, а также сбору тысяч кишечных бактерий и более совершенным методам культивирования в настоящее время доступны огромные ресурсы, позволяющие идентифицировать конкретные кишечные бактерии и их ферменты, опосредующие биотрансформацию лекарств. Однако для большинства описанных в литературе восстановительных метаболитов ответственные кишечные бактерии и их ферменты, опосредующие эти реакции, в значительной степени неизвестны. Идентификация и характеристика этих кишечных бактерий и их ферментов не только заложит основу для интеграции кишечной микробиоты в оптимизацию результатов лекарственной терапии у хозяина, но и даст возможность определить их физиологическую роль в бактериях и в кишечном сообществе [11]. 2. ОПОСРЕДОВАННОЕ ВЛИЯНИЕ МИКРОБИОТЫ НА ЛЕКАРСТВЕННУЮ ТЕРАПИЮ Отдельный интерес представляет влияние микробиоты на противоопухолевые препараты. На примере колоректального рака были выяснены некоторые важные нюансы: во-первых, бактерии играют неравноценную роль в развитии опухоли и взаимодействуют между собой (концепция «водитель-пассажир»: одни бактерии способствуют развитию опухоли, а другие селятся на ней, как на комфортном для них месте обитания), во-вторых, по мере развития опухоли изменяется и состав локального сообщества, и субстрат, на котором они находятся, поскольку микроорганизмы модифицируют свой локальный биотоп; в-третьих, бактерии в опухоли распределены неравномерно [12], что может оказывать локальное влияние на метаболические процессы. При этом результаты локальных влияний могут комбинироваться из-за того, что эти локальные микросистемы не изолированы друг от друга. Кроме того, сочетание метаболитов нескольких бактерий может приводить к иным вариантам биотрансформации по сравнению с биотрансформацией метаболитами этих же бактерий по отдельности. Для изучения неоднородности распределения отдельных видов бактерий может быть применена, например, технология HiPR-FISH, позволяющая идентифицировать и в высоком разрешении картировать пространственное распределение микроорганизмов в сложном сообществе [13]. Ранее было замечено [14], что культивирование in vitro двух типов опухолевых клеток человека вместе с фибробластами привела к неожиданному выживанию опухолевых клеток после воздействия химиотерапевтическим препаратом гемцитабином. Geller и ее коллеги провели работу по исследованию этого феномена. Используя анализ ДНК-последовательностей, они обнаружили, что образец фибробластов был заражён бактерией Mycoplasma hyorhinis [15]. Сообщалось и о других примерах, когда бактерии модулировали эффект противоопухолевой терапии. Например, у пациентов с раком толстой кишки часто имеется большое количество бактерий Fusobacterium nucleatum, ассоциированных с опухолями, и это связано с худшим клиническим прогнозом [16]. В клетках колоректального рака эта бактерия может активировать пути, придающие устойчивость опухоли к препаратам фторурацил и оксалиплатин [17]. Стоит отметить, что кишечная микробиота может влиять и на реакцию организма хозяина, и на препарат и его токсичность, модулируя молекулярные пути хозяина, ответственные за метаболизм и транспорт лекарственных веществ. Такое влияние может осуществляться через воздействие на экспрессию генов хозяина, участвующих в транспорте и метаболизме лекарств, а также через вмешательство в ферментативную активность хозяина и модуляцию иммунных реакций [18, 19]. Подобное воздействие может быть опосредовано микробными метаболитами или модификацией метаболитов хозяина, в том числе - их прямой биотрансформацией после выделения их с желчью из печени. Выделение метаболитов препарата с желчью предоставляет возможность для повторной биотрансформации их ферментами β-глюкуронидазами, которые могут реактивировать препарат в кишечнике, вызывая его повышенную токсичность. Примером такого явления служит иринотекан (CPT-11), широко используемый внутривенный пролекарственный препарат для лечения рака толстой кишки. После введения иринотекана происходит сложный ряд биотрансформационных реакций. Так в результате гидролиза иринотекана карбоксилэстеразой образуется его активный метаболит SN-38, который проявляет токсичность по отношению к эпителиальным клеткам кишечника и, как полагают исследователи, усугубляет диарею, наблюдаемую практически у 80% пациентов [20]. Аналогичный механизм способствует возникновению побочных эффектов при приеме нестероидных противовоспалительных препаратов, включая диклофенак, индометацин и кетопрофен [21]. Сравнение безмикробных и колонизированных мышей показало, что микробиота влияет на экспрессию ряда генов хозяина, участвующих в метаболизме и транспортировке лекарственных веществ [18]. Это влияние на экспрессию генов хозяина микробиотой кишечника может быть локальным [22] (в тканях кишечника) или опосредованным, что включает в себя поражение печени как органа, вовлеченного в метаболизм медикаментов [23]. Сравнение безмикробных и колонизированных мышей показало, что микробиота кишечника не только меняет спектр эндогенных метаболитов (10% общих метаболитов количественно отличаются не менее, чем на 50%), но и вносит свой вклад в циркулирующие в системе кровообращения уникальные микробные соединения [24, 25]. Некоторые из этих микробных метаболитов обрабатываются хозяином так же, как и ксенобиотики [24]. Такое совпадение ответных реакций организма хозяина на лекарства и микробные метаболиты может иметь последствия в ходе медикаментозной терапии; например, оно может привести к повышению токсичности или продолжительности полураспада ксенобиотиков в результате конкуренции между лекарством и микробными метаболитами за те же ферменты хозяина, которые участвуют в детоксикации или элиминации лекарств. Лекарственные препараты в организме проходят сложный путь, связанный с перераспределением и метаболическими превращениями, и в конечном итоге - выделением. Перед входом в систему кровообращения и попаданием в целевую ткань или орган, перорально поступившие вещества подвергаются метаболизму в кишечнике и печени, что снижает возможную системную концентрацию лекарственных средств и, как следствие, их эффективность. В то же время другой показатель – биодоступность препарата – зависит уже в том числе от активности ферментов в кишечнике и печени [26]. А активность этих ферментов может варьировать между индивидуумами до десяти раз [27], что подчёркивает значительные межиндивидуальные различия в метаболизме микроорганизмов кишечника, которые могут способствовать вариациям в эффективности препарата [28]. При оценке процессов взаимодействия микробиома и лекарственных препаратов в организме следует иметь в виду выраженные различия по фармакологическим группам. Влияние микробиоты кишечника на противоопухолевую терапию может быть опосредованно через такие механизмы, как общая иммуномодуляция, увеличение числа клеток, специфически реагирующих на антигены как микробного, так и опухолевого происхождения, метаболизм, деградация (утилизация) лекарственных препаратов. В настоящее время микробиота кишечника рассматривается как дополнительный, но немаловажный целевой объект изучения эффективности противоопухолевой терапии и снижения её токсичности, а также в качестве предиктора успешности иммунотерапии [29]. Взаимовлияние применяемой иммунотерапии и микробиоты кишечника может приводить к возникновению вторичной устойчивости к противоопухолевой терапии, либо наоборот – усилению противоопухолевого эффекта. В основе сложных взаимоотношений между иммунной системой хозяина, течением опухолевого процесса в соответствии с биологией конкретного вида опухоли, комменсальной и опухолевой микробиотой и терапевтическими мероприятиями, лежат разнообразные молекулярные механизмы, привлекающие пристальное внимание исследователей. Кроме того, инструменты и методы, используемые для характеристики микробиома, постоянно развиваются, чтобы предоставлять больше данных при меньших затратах. Новые подходы в иммунотерапии злокачественных новообразований обозначили наличие связи между микробиомом кишечника и успешностью терапевтических мероприятий. Выявление точных молекулярных механизмов, с помощью которых микробиота опосредует реакцию того или иного вида опухоли на иммуно- и химиотерапию, позволит широко использовать средства модуляции микробиома с целью повышения терапевтического эффекта и снижения токсичности противоопухолевых препаратов. В частности, анти-PD1 иммунотерапия, направленная на белок 1 программируемой клеточной смерти (programmed cell death protein 1, PD1), продемонстрировала большой потенциал в лечении злокачественных новообразований, однако её успех зависит от множества факторов, одним из которых является состав микробиоты кишечника. Исследование особенностей микробиоты кишечника пациентов с меланомой показало существование взаимосвязи между микробиомом кишечника и ответом на иммунотерапию против PD-1 у этих пациентов [29-31]. Пациенты с благоприятным микробиомом кишечника могут иметь усиленные системные и противоопухолевые иммунные ответы, опосредованные повышенной презентацией антигена и улучшенной функцией эффекторных Т-клеток в микроокружении опухоли. В недавнем исследовании, выполненном на российской когорте пациентов, удалось обнаружить маркёрные микроорганизмы, ассоциированные с ответом на анти-PD1 терапию, в том числе, аналогичные обнаруженным ранее [31]. Как показал анализ, в целом позитивное влияние на успех анти-PD1 терапии оказывает скорее общая, более “здоровая” композиция микробиома, способствующая лучшему пищеварению и усвоению из пищи питательных веществ, в частности - растительного происхождения. Терапевтический потенциал модуляции микробиома кишечника у пациентов, получающих иммунотерапию с блокадой контрольных точек, может иметь немаловажное значение для успеха лечения и прогноза течения опухолевого процесса, что потребует оперативной оценки микробиома у онкологических больных в ходе терапии. Говоря о влиянии микробиоты на метаболизм, необходимо учитывать, что ксенобиотики сами могут воздействовать на кишечную микробиоту. Известно как минимум 30 лекарственных препаратов, которые непосредственно воздействуют на микробиотную активность в кишечнике [32]. Такие лекарственные средства могут индуцировать изменения в экспрессии генов у представителей нескольких таксономических групп микробиома [33], что, в свою очередь, может приводить к снижению продукции ферментов необходимых для биотрансформации. Помимо хорошо известного действия антибиотиков на микробиоту, существуют убедительные доказательства того, что воздействие таких ксенобиотиков, как тяжелые металлы, пестициды, наночастицы, полициклические ароматические углеводороды, диоксины, фураны, полихлорированные дифенилы и «некалорийные» искусственные подсластители влияют на микробиом кишечника и это, вероятно, способствует развитию метаболических, злокачественных, воспалительных или иммунных заболеваний [34]. Причём такие ксенобиотики могут попадать как непосредственно из внешней среды (например, при дыхании), так и вследствие некачественной очистки сырья, которое было использовано для производства продуктов питания или медикаментов, потребляемых человеком. Известно, что структура микробиома тесно связана с диетой [35], возрастом [36] и генетикой самого хозяина [37]. В частности, были выявлены корреляции между наличием или отсутствием отдельных таксонов бактерий и диетой, а также ассоциации, опосредованные кишечной микробиотой, между диетой и циркулирующими метаболитами у женщин с низкой минеральной плотностью костей [38]. Приведём другой пример взаимосвязи диеты, микробиоты и циркулирующих метаболитов. Сердечный гликозид дигоксин инактивируется “сердечной гликозидредуктазой” (cgr) Eggerthella lenta. Эта нежелательная инактивация может быть снижена за счет увеличения количества аргинина в пище, что свидетельствует о влиянии диетических вмешательств на взаимодействие препарата с микробиотой [39]. Только штаммы E. lenta, несущие "сердечную гликозидредуктазу", осуществляют эту биотрансформацию. Показано, что обилие сердечной гликозидредуктазы в образцах стула позволяет предсказать инактивацию дигоксина и, как следствие, снижение активности лекарственных средств. Этот пример наглядно демонстрирует, что геномный и транскриптомный анализы микробиоты кишечника могут быть полезны для прогнозирования лекарственных реакций. Все эти данные свидетельствуют о необходимости реализации комплексного подхода. 3. ВОЗМОЖНОСТИ МОДЕЛИРОВАНИЯ МЕТАБОЛИЗМА ЛЕКАРСТВЕННЫХ СОЕДИНЕНИЙ Учитывая широкий спектр лекарств, метаболизируемых кишечной микробиотой, для развития персонализированного подхода в медицине крайне важно изучить как можно более полно и различные препараты. Это особенно важно в связи с тем, что метаболические пути лекарственных средств часто совпадают. Это позволяет построить вычислительные алгоритмы, позволяющие предсказать модификации лекарственных препаратов, которые будут произведены микробиотой конкретного пациента. В работе [40] авторы построили подобную вычислительную систему, названную microbeFDT. Сначала они оценили степень химического сходства между 10822 различными веществами, включающими компоненты пищевых продуктов, лекарств и эндогенных соединений. Сходство соединений рассчитывалось на основе подсчета общих подструктур. Затем авторы использовали данные проекта Integrative Human Microbiome Project (iHMP) [41] для определения известных ферментов, участвующих в метаболизме рассматриваемых химических соединений, а также бактерий, в геноме которых содержатся гены данных ферментов. Для последующего анализа был построен граф (рис. 2), содержащий следующую информацию: 1) сходство химических структур лекарственных препаратов, 2) описанные для них побочные эффекты, 3) ферменты, участвующие в метаболических превращениях данных веществ. Это позволило предсказывать метаболизм неизвестных соединений в том случае, когда известно схожее вещество с известным метаболизмом, осуществляемым микробами.
В качестве демонстрации применимости подхода, авторами был проанализирован и экспериментально проверен микробный метаболизм противоопухолевого препарата альтретамин, применяемого при лечении рака яичников. Примерно у половины пациентов, принимавших альтретамин перорально, были выявлены различные токсические проявления со стороны желудочно-кишечного тракта. При помощи системы microbeFDT был обнаружен структурно сходный к альтретамину препарат - меламин, для которого имелось экспериментальное подтверждение токсичности, опосредованной микробиотой. Также, опираясь на построенный граф, был найден фермент (N-деметилаза), вероятно, осуществляющий деметилирование данного соединения. Инкубирование альтретамина с фекальной суспензией и анализ при помощи LC-MS подтвердил наличие данной модификации. Воздействие на микробное сообщество (уничтожение живых микроорганизмов) значительно снизило уровень наблюдаемой модификации. Отсутствие реалистичных механистических моделей существенно тормозит понимание неоднородности реакций человека на фармакологические препараты. Хотя фармакокинетические модели подробно описывают распределение в организме и метаболизм лекарственного средства, они не учитывают индивидуальные вариации микробиома в дополнение к генетическим вариациям. Последние достижения в области создания микрофлюидных систем могут обеспечить новые подходы к анализу лекарственных соединений in vitro, включая их скрининг, активное тестирование и изучение метаболизма [42]. На сегодняшний день функции различных органов и тканей, таких как печень [42], почки [43], легкие и кишечник, воспроизводятся в виде in vitro моделей формате 2D и 3D (сфероиды, органоиды) клеточных культур. Со-культивирование нескольких типов функциональных клеток в микрофлюидных системах типа “орган-на-чипе” может рассматриваться как новая вариация скрининговых платформ ADME (всасывание, метаболизм, распределение и выведение), позволяющая одновременно и комплексно анализировать клетки, лекарственные средства и их метаболиты. Для прогнозирования сложных органных взаимодействий предложена система "тело-на-чипе", интегрирующая в себя функции нескольких органов на едином микрофлюидном устройстве [44, 45]. Особенно перспективным такой подход выглядит в отношении изучения онкологических заболеваний, поскольку он позволяет усовершенствовать разработку средств таргетной терапии [46-48]. Максимальное приближение модели биотрансформации лекарственных соединений к условиям in vivo невозможно без внесения в подобные устройства компонентов микробиома. В течение многих лет исследователи разрабатывают системы моделирования микробиоты кишечника in vitro с помощью различных типов биореакторов, а также с использованием клеточных субстратов, которые призваны имитировать клеточное разнообразие, присутствующее в кишечнике человека, поток метаболитов и перистальтические движения. Среди биореакторов, позволяющих вести непрерывные культуры, различают хемостаты, ауксостаты и ретентостаты. Все они состоят из резервуаров, датчиков, системы для ввода питательной среды и выведения продуктов жизнедеятельности для поддержания стабильных условий, однако способ создания стабильности отличается. В хемостатах удельная скорость роста регулируется за счет регулирования скорости добавления свежей среды и выведения продуктов жизнедеятельности и части клеток. В ауксостатах поддерживается стабильная плотность популяции [49]. В ретентостатах, являющихся модификацией хемостатов, происходит отток сточных вод, а биомасса задерживается с помощью специальных фильтров. Стабильность в ретентостатах поддерживается за счет ограниченности пищевого ресурса. Поэтому темпы прироста микроорганизмов в них близки к нулю [50]. Также описаны механо-хемостаты — микрофлюидные устройства, который позволяет изучать влияние механического напряжения на рост микроорганизмов [51]. Недавно описан прибор «Омнистат» (Omnistat), который, в зависимости от настроек (температура, pH, оксигенация, приток свежей среды и т.д.), может работать и как хемостат, и как аукостат, и как ретентостат. В этой системе предусмотрена возможность потоков между сосудами, работающими в разных режимах и поддерживающих разные условия культивирования [52]. Очевидно, что адекватное моделирования микробиома кишечника in vitro требует создания моделей с использованием технологий клеточной биологии, среди которых наиболее перспективными можно считать органоиды кишечника. Органоиды создаются из индуцированных стволовых клеток, которые при определенных условиях культивирования пролиферируют и образуют сферы. В этих сферах присутствует кишечный эпителий, мезенхима и структуры, подобные кишечному просвету [53]. Органоиды как модель используются для широкого спектра исследований — от взаимодействия тканей кишечника с микробиомом [54] до изучения вирусных [55] и паразитарных инфекций. Более продвинутая технология, подобная органоидам, — создание модели органа на искусственном каркасе-матрице [56]. Такие модели могут создаваться из различных клеточных линий, в том числе, в комбинации, а также позволяют создавать гибридные органы [57]. По своей сути, данная технология представляет универсальное решение для любых исследовательских задач и ограничивается лишь сложностью каркаса-матрицы. Как уже отмечалось выше, в настоящее время активное развитие получили всевозможные микрофлюидные устройства, реализующие системы “орган-на-чипе” [58]. В настоящее время существует ряд таких устройств [59], однако, особое внимание привлекает микрофлюидное устройство HuMiX [60, 61]. Устройство разработано для изучения взаимодействия микробиоты и желудочно-кишечного тракта человека и состоит из трех отсеков, разделённых полупроницаемыми мембранами, что позволяет одновременно культивировать как эукариотические клетки, так и микробные штаммы. Другие устройства позволяют совместно культивировать сложные сообщества анаэробных и аэробных комменсальных бактерий и кишечный эпителий [62]. Сегодня мы еще далеки от создания надежных микрофлюидных устройств типа “тело-на-чипе”, реализующих гетерологичные экосистемы, аналогичные микробиому кишечника. Однако, создание таких устройств, начиная с моделей кишечника in vitro, крайне важно для перехода к высокопроизводительной проверке влияния микроорганизмов кишечника на широкий спектр ксенобиотиков (и наоборот), включая детальный анализ структурно-функциональных свойств и построение предсказательных вычислительных моделей. ЗАКЛЮЧЕНИЕ В настоящее время вопрос о влияния микробиоты на метаболизм лекарственных средств остается недостаточно изученным, однако существуют предпосылки для прорыва в этой области. Исходя из вышеописанного, можно заключить, что изучение влияния микробиоты на эффективность препаратов требует комплексного подхода. Разработка сложных контролируемых моделей позволит детально прояснить механизмы действия лекарственных средств, что, в свою очередь, даст возможность более взвешенно принимать решение о назначении того или иного лекарственного средства. Полученные в ходе таких исследований данные позволят не только усовершенствовать уже существующие терапевтические методы за счет выявления новых факторов, влияющих на эффективность того или иного препарата, но и разработать принципиально новые подходы к лечению. Сложные микрофлюидные системы на основе комплекса типа «тело-на-чипе» позволят проводить исследования с высоким уровнем контроля условий, с низким уровнем упрощения системы относительно живого организма и в высоком разрешении. ФИНАНСИРОВАНИЕ Работа выполнена при финансовой поддержке Министерства науки и высшего образования (соглашение №RFMEFI60419X0215). СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВ Настоящая статья не содержит каких-либо исследований с участием людей или использованием животных в качестве объектов. КОНФЛИКТ ИНТЕРЕСОВ Все авторы заявляют об отсутствии конфликта интересов. ЛИТЕРАТУРА
|