СОДЕРЖАНИЕ
ВВЕДЕНИЕ
МАТЕРИАЛЫ И МЕТОДЫ
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
ЗАКЛЮЧЕНИЕ
СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВ
ФИНАНСИРОВАНИЕ
КОНФЛИКТ ИНТЕРЕСОВ
ЛИТЕРАТУРА
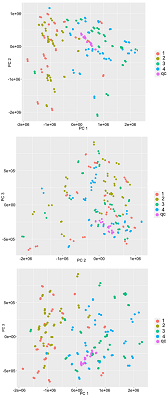
Рисунок 1Распределение биологических образцов, не подвергавшиеся нормализации в пространстве трёх главных компонент. Группа 1 – нормальная ткань молочной железы от пациентов без метастазирования, группа 2 – ткань нормальной ткани молочной железы от пациентов с метастазированием, группа 3 – опухолевая ткань молочной железы от пациентов без метастазирования, группа 4 – опухолевая ткань молочной железы от пациентов с метастазированием, группа qc – группа образцов контроля качества.
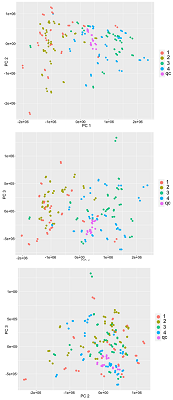
Рисунок 2Распределение биологических образцов после автомасштабирования в пространстве трёх главных компонент. Группа 1 – нормальная ткань молочной железы от пациентов без метастазирования, группа 2 – ткань нормальной ткани молочной железы от пациентов с метастазированием, группа 3 – опухолевая ткань молочной железы от пациентов без метастазирования, группа 4 – опухолевая ткань молочной железы от пациентов с метастазированием, группа qc – группа образцов контроля качества.
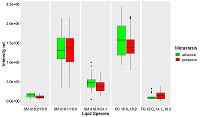
Рисунок 3Диаграмма размахов относительных уровней липидов, являющимися маркерами регионарного метастазирования в опухолевых тканях при раке молочной железы. Зелёным обозначено отсутствие метастазирования, красным – наличие.
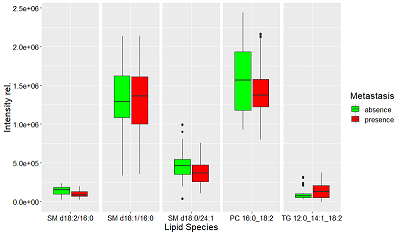
Рисунок 4Диаграмма размахов относительных уровней липидов, являющимися маркерами регионарного метастазирования в тканях молочной железы при раке молочной железы. Зелёным обозначено отсутствие метастазирования, красным – наличие.
Таблица 1Коэффициенты при липидах-маркерах в опухолевых тканях β, их доверительный интервал (ДИ β), критерий Вальда и вероятность отличия коэффициента от нуля (p).
Таблица 2. Коэффициенты при липидах-маркерах в здоровых тканях β, их доверительный интервал (ДИ β), критерий Вальда и вероятность отличия коэффициента от нуля (p).
|
Алгоритм обработки масс-спектрометрических данных для получения диагностической панели молекулярных соединений на примере поиска маркеров метастазирования при раке молочной железы
А.О. Токарева1,2, В.В. Чаговец3, А.С. Кононихин1,3, Н.Л. Стародубцева2,3,
В.Е. Франкевич3, Е.Н. Николаев1*
Ключевые слова: масс-спектрометрия; обработка данных; биологические маркеры
Использование молекулярных маркеров является одним из способов диагностики заболевания. Масс-спектрометрия (МС) является чувствительным методом молекулярного анализа, который позволяет эффективно производить как целевой, так и нецелевой анализ образца, что делает его эффективным для поиска потенциальных маркеров заболевания в полученном молекулярном профиле образца. Использование предварительного хроматографического разделения увеличивает число соединений, регистрируемых масс-спектрометром в образце, и повышает точность идентификации соединений. Липиды – группа амфифильных соединений низкой молекулярной массы ( меньше 2000 Да), участвующая в построении мембран клеток, передаче внутриклеточных и межклеточных сигналов и транспорте, запасании энергии и др. Уже показана взаимосвязь уровней отдельных липидов и классов липидов с наличием заболевания: ростом уровня фосфатидилхолинов при раке [1], падением уровня сфингомиелинов при болезни Альцгеймера [2]. Также в ряде работ получены панели липидов-маркеров для диагностики заболевания [3–5].
В данной работе предлагается последовательность действий для обработки данных, полученных с использованием хромато-масс-спектрометрического анализа для создания диагностической панели маркеров на примере диагностики метастазирования в регионарные лимфоузлы при раке молочной железы
В работе использовали образцы ткани от 40 пациентов с раком молочной железы без регионарного метастазирования (средний возраст 56.3 года, у 20 пациентов стадия I, у 20 – стадия II. Рак у 20 пациентов относился по классификации TNM относился к группе T1N0M0, у 20 – к группе T2N0M0) и от 48 пациентов с раком молочной железы с регионарным метастазированием (средний возраст пациентов составил 56.7 лет, у 32 пациентов была II стадия рака, у 16 – III стадия рака, Рак у 10 пациенток по классификации TNM относился к группе T1N1M0, у 36 – к группе T2N1-3M0, у 2 – к группе T3N3M0): биопсийные материалы опухолевой ткани молочной железы и нормальной ткани молочной железы. Таким образом, пул образцов состоял из 4 клинических групп:
- Группа 1 – нормальная ткань молочной железы от пациентов без метастазирования;
- Группа 2 – ткань нормальной ткани молочной железы от пациентов с метастазированием;
- Группа 3 – опухолевая ткань молочной железы от пациентов без метастазирования;
- Группа 4 – опухолевая ткань молочной железы от пациентов с метастазированием.
Из образца тканей весом 40 г методом Фолча[6] выделяли липиды с последующим перерастворением в 200 мкл изопропанол/ацетонитрил 1/1. На основе 10 мкл. от каждого экстракта был создан образец контроля качества. Полученные липидные экстракты (без образца контроля качества) были разбиты на 3 партии для анализа:
- партия 1: 16 образцов из группы 1, 14 образцов из группы 2, 16 образцов из группы 3, 14 образцов из группы 4;
- партия 2: 10 образцов из группы 1, 20 образцов из группы 2, 10 образцов из группы 3, 20 образцов из группы 4;
- партия 3: 14 образцов из группы 1, 14 образцов из группы 2, 14 образцов из группы 3, 14 образцов из группы 4.
Разделение липидных экстрактов осуществляли на хроматографе Dionex UltiMate 3000 («Thermo Scientific», Германия) с использованием обратно-фазовой колонки Zorbax C18 (длина 150 мм, внутренний диаметр 2.1 мм, размер частиц 5 мкм, «Agilent», США) и следующих элюентов в качестве подвижной фазы: элюент А - ацетонитрил/вода (60/40, о/о) с добавлением 0,1% муравьиной кислоты и 10 мМ формиата аммония; элюент В - ацетонитрил/изопропанол/вода, (90/8/2, о/о/о), с добавлением 0,1% муравьиной кислоты и 10 мМ формиата аммония. Скорость потока 35 мкл/мин, температура колонки 50оС. Доля градиента В изменялась по заданному алгоритму: 0-0.5 мин – 30% В, до 20-ой минуты росла до 99% и сохраняла значение до 30-ой минуты и за полминуты возвращалось к значению 30%. МС анализ производили с использованием прибора Maxis Impact («Bruker», Германия) со следующими настройками: диапазон 100-1800 m/z, с напряжением на капилляре 4.1 кВ в режиме положительных ионов, давлением распыляющего газа 0.7 бар, скорости потока осушающего газа 6 л/мин и температурой 200оС.
Выполнение тандемного МС анализа осуществляли с использованием зависимого сканирования, в котором после снятия спектра снимали спектры фрагментации при энергии столкновения в 35 эВ соединений, давших пять самых интенсивных пиков в спектре, с окном изоляции 5 Да и временем исключения 2 мин. Анализ образцов контроля качества производился через каждые 10 исследуемых образцов. Данные, полученные в ходе анализа в виде .d файлов, преобразовывали в формат MzXml посредством программного обеспечения msConvert (Proteowizard, 3.0.9987) и предобрабатывали с использованием алгоритма, предоставленного Koelmel [7], программного обеспечения MzMine [8]. Идентификацию липидов осуществляли при помощи программы Lipid Match [7]. Номенклатура ионов использована согласно Lipid Maps терминологии в сокращённой форме записи [9].
Обработку данных осуществляли посредством расчета для каждой партии: значения средней величины интенсивности пика каждого соединения
| $$\langle I_{p,b} \rangle={{\sum^{N_b}_{i=1}{I_{p,i,b}}}\over {N_b}}$$
|
(1)
|
и стандартного отклонения интенсивности пика каждого соединения
| $$sd\left(I_{p,b}\right)=\sqrt{{{\sum^{N_b}_{i=1}{{\left(I_{p,i,b}- \langle I_{p,b} \rangle \right)}^2}}\over {N_b-1}}}$$
|
(2),
|
где Nb – число образцов в партии b, Ip,i,b – интенсивность пика p в образце i партии b; для всего набора данных рассчитывали значения средней величины интенсивности пика каждого соединения
| $$\langle I_p \rangle ={{\sum^N_{i=1}{I_{p,i}}}\over {N}}$$
|
(3)
|
и стандартного отклонения интенсивности пика каждого соединения
| $$sd\left(I_p\right)=\sqrt{{{\sum^N_{i=1}{{\left(I_{p,i}- \langle I_p \rangle \right)}^2}}\over {N-1}}}$$
|
(4),
|
где N – общее число образцов I p,i – интенсивность пика p в образце i.
На основе этих значений рассчитывали новые значения каждого пика в каждом образце по формуле
| $$sd\left(I_p\right)=\sqrt{{{\sum^N_{i=1}{{\left(I_{p,i}- \langle I_p \rangle \right)}^2}}\over {N-1}}}$$
|
(5),
|
т.е. осуществляли автомасштабирование полученных данных.
После выполнения нормировки набор данных был разбит на два: содержащий информацию об экстрактах здоровых тканей и содержащий информацию о липидных экстрактах опухолевых тканей. Для каждого набора данных было проделано вычисление значений проекций переменной с использованием метода ортогональных проекций на скрытые структуры [10, 11]:
- выполнено парето-масштабирование матрицы независимых переменных N*m X, где N – число образцов, m – число соединений
| $$X_i={{X_i-\left\langle X_i\right\rangle }\over {\sqrt{sd(X_i)}}} $$
|
(6),
|
где Xi– i-ый столбец матрицы X, < Xi >– среднее значение i-того столбца и sd( Xi) – стандартное отклонение переменных в i-ом столбце;
- выполнено парето-масштабирование столбца зависимых переменных y высотой N, где 0 обозначается состояние «отсутствие метастазирование», 1 – состояние «болезнь»
| $$y={{y-\left\langle y\right\rangle }\over {\sqrt{sd\left(y\right)}}}$$
|
(7),
|
где <y>– среднее значение переменных отклика, sd(y) – стандартное отклонение переменных отклика;
- рассчитаны веса для независимых переменных
| $$w={{X^Ty}\over {y^Ty}} $$
|
(8);
|
- выполнена нормализация рассчитанного вектора
| $$w={{w}\over {\left\|w\right\|}} $$
|
(9);
|
- рассчитаны предсказательные счета
- рассчитана предсказательная нагрузка независимых переменных
| $$p^T=\ {{t^TX}\over {t^Tt}}$$
|
(11);
|
- вычислен вектор ортогональных нагрузок
- выполнена нормализация рассчитанного вектора ортогональных нагрузок
| $$w_o={{w_o}\over {\left\|w_o\right\|}} $$
|
(13);
|
- рассчитаны ортогональные счета
- рассчитана ортогональная нагрузка независимых переменных
| $${p_o}^T=\ {{{t_o}^TX}\over {{t_o}^Tt_o}}$$
|
(15);
|
- вычислены данные, не содержащие ортогональной составляющей
| $$X_p=X{\rm -}{t_o}^Tp_o$$
|
(16);
|
- вычислены предсказательные счета от независимых переменных, не содержащих ортогональной составляющей
- вычислена предсказательная нагрузка от независимых переменных, не содержащих ортогональной составляющей
| $${p_p}^T=\ {{{t_p}^TX_p}\over {{t_p}^Tt_p}} $$
|
(18);
|
- нормирован вектор, содержащий предсказательную нагрузку от независимых переменных без ортогональной составляющей
| $$p_p={{p_p}\over {\left\|p_p\right\|}} $$
|
(19);
|
- рассчитан вектор, содержащий значения проекций переменной
| $$VIP=p_p\sqrt{m} $$
|
(20).
|
Полученные значения проекции переменной (ПП) были использованы в качества критерия выбора соединений-потенциальных маркеров, где нижней границей значения проекции переменной для маркера принималась 1.
Далее произвольно была выбрана переменная из набора переменных, сформированного на основе значений ПП. После этого следует этап расчёта информационного критерия Акаике (ИКА):
- выполняем поиск коэффициентов для логистической регрессии, где независимой переменной является выбранная выше переменная, переменной отклика – наличие или отсутствие метастазирование (1 или 0 соответственно) методом максимизации функции правдоподобия
| $$l=\sum^M_{i=1}{y_i\left(β^Tx_i\right)-ln\left(1+e^{β^Tx_i}\right)} $$
|
(21),
|
где y i – переменная отклика, принимающая значения 0 или 1, x i – объединённый вектор единицы и независимых переменных, β – объединённый вектор свободного члена и коэффициентов при переменных, M – число задействованных переменных
- с использованием вычисленных коэффициентов вычисляем лог-функцию правдоподобия и ИКА
- повторяем п.п. 1 – 3 для всех m переменных.
- выбрираем переменную, для которой рассчитанное значение AIC будет максимальным (обозначим это значение как AIC’);
- выполняем п.п. 1 – 5 для комбинации «выбранная ранее переменная + каждая из оставшихся переменных»;
- сравниваем AIC со значением AIC’;
- если AIC из п. 7 больше AIC’, повторяем п.п. 1-7, имея в качестве постоянных переменных переменные, отобранные ранее и обозначив как AIC’ значение из п. 7; Если AIC из п. 7 меньше AIC’, п. 9;
- переменные, при которых было получено AIC’ и рассчитанные для них коэффициенты используем дальше.
Далее осуществляли проверку на статистически значимое неравенство коэффициентов при переменных нулю с удалением переменных, не удовлетворявших этому условию:
- для отобранных ранее переменных вычисляли их коэффициенты в логистической регрессией на основе максимизации функции правдоподобия;
- подставив вычисленные значения β в матрицу
| $$D{\rm =}\sum^n_{i=1}{{{x_i{x_i}^Te^{{β^Tx}_i}}\over {{\left(1+e^{{β^Tx}_i}\right)}^2}}}$$
|
(23),
|
вычисляли значения стандартной ошибки для коэффициента βj
| $${\rm SE}\left(β^j\right)=\sqrt{D_{jj}}$$
|
(24),
|
где j – порядковый номер коэффициента в векторе β;
- вычисляли вероятность отличия от нуля коэффициента βj
| $$p_j={β\left({{β^j}\over {{\rm SE}\left(β^j\right)}}\right)}^2$$
|
(25),
|
где χ(θ) 2– распределение квадрата независимой стандартной нормальной случайной величины θ;
- если pj > 0.05, то переменная, соответствующая pj и не являющаяся 1, исключается из задействованного набора переменных и действия 1- 4 повторяются.
Качество построенных моделей проверяли с использованием скользящего контроля по отдельным объектам, в котором модель тренировалась на N-1 объекте и тестировалась на оставшемся объекте N раз.
Изменение распределения координат образцов в первых трёх координатах главных компонент в результате нормализации представлены на рисунках 1 и 2. После нормализации относительное отклонение значения полного ионного тока для образцов контроля качества снизилось с 7% до 4%.

|
Рисунок 1.
Распределение биологических образцов, не подвергавшиеся нормализации в пространстве трёх главных компонент. Группа 1 – нормальная ткань молочной железы от пациентов без метастазирования, группа 2 – ткань нормальной ткани молочной железы от пациентов с метастазированием, группа 3 – опухолевая ткань молочной железы от пациентов без метастазирования, группа 4 – опухолевая ткань молочной железы от пациентов с метастазированием, группа qc – группа образцов контроля качества.
|

|
Рисунок 2.
Распределение биологических образцов после автомасштабирования в пространстве трёх главных компонент. Группа 1 – нормальная ткань молочной железы от пациентов без метастазирования, группа 2 – ткань нормальной ткани молочной железы от пациентов с метастазированием, группа 3 – опухолевая ткань молочной железы от пациентов без метастазирования, группа 4 – опухолевая ткань молочной железы от пациентов с метастазированием, группа qc – группа образцов контроля качества.
|
В случае опухолевой ткани молочной железы для 54 из 317 идентифицированных соединений значение ПП составило больше 1. Эти соединения преимущественно относятся к классам триацилглицеридов (31), фосфатидилхолинов (14), сфингомиелинов (6) и диацилглицеридов (3).
В случае нормальной ткани молочной железы для 60 из 317 идентифицированных соединений значение ПП больше оказалось больше 1. Данные соединения относятся к классам лизо- и фосфатидилхолинов (20), триацилглицеридов (18), диагцилглицеридов (12), сфингомиелинов (6), фосфатидилэтаноламинов (4).
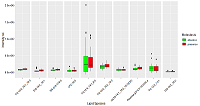
После выбора переменных по значению ИКА и удалению переменных со статистически незначимо отличающимися от ноля коэффициентами для диагностики наличия регионарного метастазирования по опухолевой ткани получаем набор липидов {SM 18:2/16:0, SM 18:1/16:0, SM 18:0/24:1, PC 16:0_18:2, TG 12:0_14:1_18:2} (таблица 1, рисунок 3). Тестирование модели дало значение площади под операционной кривой 0.81, чувствительность и специфичность 94% и 65% при пороге 0.39.
|
Закрыть окно

|
Таблица 1.
Коэффициенты при липидах-маркерах в опухолевых тканях β, их доверительный интервал (ДИ β), критерий Вальда и вероятность отличия коэффициента от нуля.
|
|
Рисунок 3.
Диаграмма размахов относительных уровней липидов, являющимися маркерами регионарного метастазирования в опухолевых тканях при раке молочной железы. Зелёным обозначено отсутствие метастазирования, красным – наличие.
|
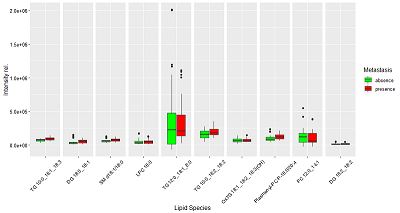
После выбора переменных по значению ИКА и удалению переменных со статистически незначимо отличающимися от ноля коэффициентами для диагностики наличия регионарного метастазирования по биопсии ткани молочной железы получаем набор липидов {TG 10:0_18:1_18:3, DG 18:0_18:1, SM d18:1/18:0, LPC 16:0, TG 12:0_18:1_8:0, TG 10:0_18:2_18:2, OxTG 18:1_18:2_18:3(OH), PC P-16:0/20:4, PC 12:0_14:1, DG 18:2_18:2} (табл. 2, рис. 4). Тестирование модели дало значение площади под операционной кривой 0.79, чувствительность и специфичность 88% и 58% при пороге 0.15.
|
Закрыть окно

|
Таблица 2.
Коэффициенты при липидах-маркерах в здоровых тканях β, их доверительный интервал (ДИ β), критерий Вальда и вероятность отличия коэффициента от нуля.
|
|
Рисунок 4.
Диаграмма размахов относительных уровней липидов, являющимися маркерами регионарного метастазирования в опухолевых тканях при раке молочной железы. Зелёным обозначено отсутствие метастазирования, красным – наличие.
|
Полученные модели характеризуются высокой чувствительностью. В модели, построенной для диагностики метастазирования по биопсии опухолевой ткани, в качестве маркеров преобладают сфигномиелины (3 из 5), в модели, построенной для диагностики по биопсии ткани молочной железы, преобладают триглицериды (4 из 10) и фосфатидилхолины (3 из 10). Уже показано, что сфингомиелины являются маркерами метастатических процессов [13, 14]. Триглицериды являются основным источником энергии в клетках [15]. Фосфатидилхолины связаны с пролиферативными процессами [16].
Предложенный метод дал возможность построить диагностическую панель липидов для моделей высокой чувствительности. Наличие в моделях липидов, для чьих классов определена связь с метастатическими и опухолеобразующими процессами, говорит о релевантности предложенного метода обработки данных.
Все клинические исследования проводили в соответствии с принципами, изложенными в Хельсинкской декларации. Все пациенты прочитали и подписали информированное согласие, одобренное этическим комитетом Национального медицинского исследовательского центра акушерства, гинекологии и перинатологии имени ак. В.И. Кулакова (протокол No9 от 22.11.2018).
Работа выполнена при поддержке Мегагранта Министерства науки и высшего образования Российской Федерации (Соглашение со Сколковским институтом науки и технологий, № 075-10-2019-083 от 11 декабря 2019 г.)
Авторы заявляют об отсутствии конфликта интересов.
- Podo, F., Canevari, S., Canese, R., Pisanu, M.E., Ricci, A., Iorio, E. (2011) Tumour Phospholipid Metabolism. Exp. Oncol. 19, 1–10.
- Wong, M.W., Braidy, N., Poljak, A., Pickford, R., Thambisetty, M., Sachdev, P.S. (2017) Dysregulation of lipids in Alzheimer’s disease and their role as potential biomarkers. Alzheimer’s Dement. 13(7), 810–27, DOI
- Liu, X., Li, J., Zheng, P., Zhao, X., Zhou, C., Hu, C., Hou, X., Wang, H., Xie, P., Xu, G. (2016) Plasma lipidomics reveals potential lipid markers of major depressive disorder. Anal. Bioanal. Chem. 408(23), 6497–507, DOI
- Anand, S., Barnes, J.M., Young, S.A., Garcia, D.M., Tolley, H.D., Kauwe, J.S.K., Graves, S.W. (2017) Discovery and Confirmation of Diagnostic Serum Lipid Biomarkers for Alzheimer’s Disease Using Direct Infusion Mass Spectrometry. J. Alzheimer’s Dis. 59(1), 277–90, DOI
- Hogan, S.R., Phan, J.H., Alvarado-Velez, M., Wang, M.D., Bellamkonda, R. V., Fernández, F.M., Laplaca, M.C. (2018) Discovery of Lipidome Alterations Following Traumatic Brain Injury via High-Resolution Metabolomics. J. Proteome Res. 17(6), 2131–43, DOI
- Folch, J., Lees, M., Sloane Stanley, G.H. (1957) A simple method for the isolation and purification of total lipides from animal tissues. J. Biol. Chem. 226(1), 497–509.
- Koelmel, J.P., Kroeger, N.M., Ulmer, C.Z., Bowden, J.A., Patterson, R.E., Cochran, J.A., Beecher, C.W.W., Garrett, T.J., Yost, R.A. (2017) LipidMatch: An automated workflow for rule-based lipid identification using untargeted high-resolution tandem mass spectrometry data. BMC Bioinformatics 18(1), 1–11, DOI
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. (2010) MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics 11, DOI
- Sud, M., Fahy, E., Cotter, D., Brown, A., Dennis, E.A., Glass, C.K., Merrill, A.H., Murphy, R.C., Raetz, C.R.H., Russell, D.W., Subramaniam, S. (2007) LMSD: LIPID MAPS structure database. Nucleic Acids Res. 35(SUPPL. 1), 527–32,
- Wold, S., Sjöström, M., Eriksson, L. (2001) PLS-regression: a basic tool of chemometrics. Chemom. Intell. Lab. Syst. 58(2), 109–30, DOI
- Galindo-Prieto, B., Eriksson, L., Trygg, J. (2015) Variable influence on projection (VIP) for OPLS models and its applicability in multivariate time series analysis. Chemom. Intell. Lab. Syst. 146, 297–304, DOI
- Akaike, H. (1998) Information Theory and an Extension of the Maximum Likelihood Principle. Sel. Pap. Hirotugu Akaike, 199–213, DOI
- Roy, J., Dibaeinia, P., Fan, T.M., Sinha, S., Das, A. (2019) Global analysis of osteosarcoma lipidomes reveal altered lipid profiles in metastatic versus nonmetastatic cells. J. Lipid Res. 60(2), 375–87, DOI
- Peng, W., Tan, S., Xu, Y., Wang, L., Qiu, D., Cheng, C., Lin, Y., Liu, C., Li, Z., Li, Y., Zhao, Y., Li, Q. (2018) LC-MS/MS metabolome analysis detects the changes in the lipid metabolic profiles of dMMR and pMMR cells. Oncol. Rep. 40(2), 1026–34, DOI
- Garrett, R.H., Grisham, C.. (2016) Biochemistry. 6th ed., CENGAGE Learning.
- Fagone, P., Jackowski, S. (2013) Phosphatidylcholine and the CDP-choline cycle. Biochim. Biophys. Acta - Mol. Cell Biol. Lipids 1831(3), 523–32, DOI
|








