К 40-летию Института физиологически активных веществ РАН
|
MOLTRA-II. Новые дескрипторы водородной связи Институт физиологически активных веществ Российской академии наук 142432 Черноголовка Московской обл., Северный проезд, 1; e-mail: yarkov@ipac.ac.ru Ключевые слова: 3-D дескрипторы; дескрипторы водородной связи DOI: 10.18097/BMCRM00069 ВВЕДЕНИЕ Роль водородных связей (Н-комплексов) в биологических системах хорошо известна. Ранее нами создан программный комплекс HYBOT [1], позволяющий рассчитать термодинамические параметры водородной связи по данным экспериментальных исследований водородной связи методами спектроскопии и калориметрии. На основе собранных данных удалось создать двумерные дескрипторы водородной связи, которые могут быть использованы как для предсказания энергетических параметров водородной связи, так и для включения в список дескрипторов при молекулярном моделировании структура-свойство. В работе [2] нами описана программа MOLTRA-II, созданная на основе системы управления химическими базами данных CHED [3], позволяющая учитывать пространственную структуру взаимодействующих молекул на основе расчета спектра межатомных расстояний. Описаны методы расчета энергии стерического (Van der Waals), электростатического взаимодействия атомов, а также энергии взаимодействия атомов доноров и акцепторов водородной связи. Рассчитанные на этой основе дескрипторы хорошо описывают точечные химические Н-комплексы состава 1:1. Сложные органические соединения, лекарственные препараты и, тем более, молекулы-фрагменты живых систем обладают несколькими центрами водородного связывания, и для их описания необходим подход с учетом множества водородных связей. Особенно важен учёт водородного связывания в работах in silico, не всегда придающих этому фактору должного значения. Тем не менее, многие современные процедуры трехмерного компьютерного дизайна включают расчеты силового поля для учета водородного связывания, которые используются во многих областях молекулярных исследований, таких как докинг лигандов, конформационный анализ, идентификация фармакофоров и др.[4-6]. С другой стороны, поиск простых дескрипторов, связанных как с трехмерной структурой, так и с образованием водородной связи значительно упрощает их использование в качестве обычных дескрипторов в моделировании методами анализа связи структура-активность (QSAR). Программа MOLTRA-II решает, в основном, последнюю задачу. ДЕСКРИПТОРЫ ПРОГРАММЫ MOLTRA-II В качестве одного из таких дескрипторов для расчета свойств химических соединений часто используется PSA (Polar Surface Area) [7]. PSA определяется как доля молекулярной вандерваальсововой поверхности, которая связана с атомами кислорода, азота и атомами водорода при этих атомах. Была установлена высокая корреляция PSA с числом донорных и акцепторных атомов (Na и Nd) [8]. В дальнейшем, с учетом силы водородного связывания, рассчитанной HYBOT [1], были предложены шесть HYBOT PSA дескрипторов, вычисляемых по формулам (1-6) [8,9]:
Приведенные шесть дескрипторов дают возможность определить области молекулярной поверхности, благоприятные для образования межмолекулярных водородных cвязей и учесть влияние заместителей при донорных и акцепторных центрах на силу этих связей. Расчет другого набора из четырех дескрипторов для оценки поверхностей оптимального водородного связывания (OSA-Optimal Surface Area) [8,9] также реализован в программе MOLTRA-II. Оптимальному водородному связыванию соответствует минимум потенциальной энергии водородной связи. Часть поверхности (в Å2) вокруг изучаемой молекулы, где взаимодействие акцепторных атомов с донорными атомами молекулы-пробы оптимально, пропорциональна абсолютному значению произведения энтальпийных факторов HYBOT (или факторов свободной энергии HYBOT), описывается дескриптором OSAEDprobe (или OSAСDprobe). В случае пробы – донора протонов они рассчитываются по формулам:
Часть поверхности (в Å2) вокруг изучаемой молекулы, где взаимодействие донорных атомов водорода с акцепторными атомами молекулы-пробы оптимально, пропорциональна абсолютному значению произведения энтальпийных факторов HYBOT (или факторов свободной энергии HYBOT) описывается дескриптором OSAEAprobe (или OSAСAprobe). В случае пробы – акцептора протонов они рассчитываются по формулам:
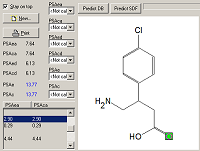
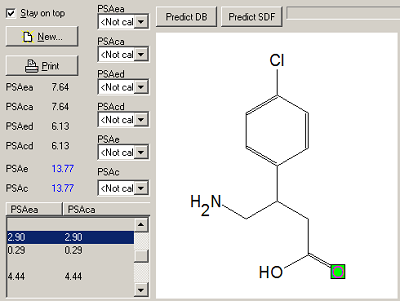
Программа MOLTRA-II рассчитывает также энтальпию и свободную энергию водородной связи двух молекул при оптимальном расположении донорных и акцепторных атомов водорода и акцептора [9]. РАСЧЕТ PSA ДЕСКРИПТОРОВ ПРОГРАММОЙ MOLTRA-II Для расчета HYBOT PSA дескрипторов в меню Tools программы MOLTRA-II добавлена опция «New hydrogen bond descriptors». Программа предоставляет данные расчета PSA как для задаваемой молекулы, так и для отдельного ее атома (рис.1).
Для расчета ОSA дескрипторов в меню Tools программы MOLTRA-II добавлена опция Interaction with probas, для расчета энергии водородной связи опция – Energy of interaction. Использование поверхностных дескрипторов водородной связи продемонстрировано нами ранее [8] при моделировании пассивной диффузии 154 лекарств.
В частности, для уравнения пассивной диффузии FA (11) с X дескриптором PSA были получены следующие коэффициенты и статистические критерии: a1 = 1.98(+/-0.19), a2 = -0.008(+/-0.001), n = 154, r = 0.86, S = 0.18, F = 413.1 Таким образом, программа MOLTRA-II предоставляет возможность расширить спектр дескрипторов при создании широкого спектра QSAR моделей, если описываемое явление связано с образованием водородных связей. Некоммерческое использование программы для расчета дескрипторов доступно при обращении к авторам. БЛАГОДАРНОСТИ Работа выполнена в рамках государственного задания на 2018 год (тема № 0090-2017-0020). ЛИТЕРАТУРА
|