К 40-летию Института физиологически активных веществ РАН
|
Расчет и анализ фрактальных дескрипторов для белковых аминокислот в различных конформационных состояниях
1Институт физиологически активных веществ Российской академии наук
142432 Черноголовка Московской обл., Северный проезд, 1;
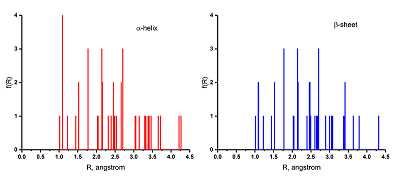
*e-mail: beng@ipac.ac.ru Ключевые слова: аминокислоты; фрактальные дескрипторы; α-спираль; β-структура DOI: 10.18097/BMCRM00070 ВВЕДЕНИЕ Одним из ключевых вопросов в молекулярной биологии является выявление связи между физиологической ролью белков и их первичной структурой. Хорошо известно, что функции белков и их пространственная структура в значительной степени определяются последовательностью аминокислотных остатков [1,2]. В качестве одного из подходов для выявления и анализа таких зависимостей используются методы нелинейной динамики и фрактальной геометрии [3-6]. В таких работах объектами изучения обычно являются полимерные молекулы (пептиды и белки). При этом достаточно широкое распространение получили исследования структуры белков, связанные с расчетом фрактальных размерностей [7-10]. Однако анализу мономеров белковых молекул уделяется мало внимания. В частности, отсутствуют данные о влиянии изменения конформации аминокислот на их фрактальные характеристики. Ранее было предложено применять для описания структуры соединений гистограммы межатомных расстояний (ГМА) и использовать их для расчета фрактальных размерностей (дескрипторов) [11]. Эти дескрипторы весьма чувствительны и количественно отражают тонкие изменения в пространственной структуре молекул. Целью настоящей работы является расчет и анализ фрактальных дескрипторов для 20 белковых аминокислот в двух конформационных состояниях: α-спираль и одноцепочечная β-структура. МАТЕРИАЛЫ И МЕТОДЫ В качестве объектов исследования использовали 20 белковых аминокислот. Трехмерную структуру молекул рассчитывали на основе базы данных аминокислот из компьютерной программы HyperChem [12]. Для анализа использовали 2 конформации: α-спираль (L; ϕ = −58°; ψ = −47°; ω = 180°) и одноцепочечную β-структуру (L; ϕ = 180°; ψ = 180°; ω = 180°). Для расчета фрактальных дескрипторов применяли гистограммы межатомных расстояний с разрешением 0.01 Å (рис. 1). По своему физическому содержанию фрактальный дескриптор представляет собой фрактальную размерность, рассчитанную для определенной группы межатомных расстояний с использованием известного клеточного алгоритма [13]. В ГМА использовали четыре части: 1) все атомы; 2) область валентно связанных атомов; 3) область атомов, находящихся в Ван-дер-Ваальсовом контакте; 4) область несвязанных атомов. В результате было рассчитано 4 типа дескрипторов: Dtot, Dval, Dvdw и Dunb соответственно (табл. 1, 2).
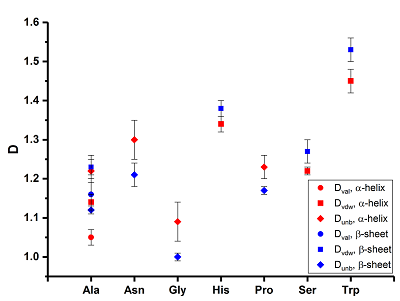
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ Фрактальная размерность является количественной мерой «изрезанности» ГМА и, теоретически, для плоской кривой может меняться от 1 до 2. Интервал изменения фрактальных дескрипторов в случае α-спирали составлял 1.17 ÷ 1.45, 1.01 ÷ 1.33, 1.11 ÷ 1.45, 1.09 ÷ 1.41 для Dtot, Dval, Dvdw, Dunb соответственно. Для β-структуры эти величины были равны 1.21 ÷ 1.45, 1.01 ÷ 1.28, 1.11 ÷ 1.53 и 1.00 ÷ 1.42.
Как следует из представленных данных, интервалы изменения дескрипторов в двух разных конформациях довольно близки и, в целом, можно ожидать близости их значений для отдельных молекул аминокислот. И, действительно, в большинстве случаев величины фрактальных дескрипторов мало отличаются друг от друга. Однако имеются и различия для ряда аминокислот (рис. 2). Это проявляется в том, что интервалы варьирования фрактальных дескрипторов не перекрываются в различных конформационных состояниях.
При этом можно отметить, что эти изменения связаны только с тремя дескрипторами: Dval, Dvdw, Dunb. Фрактальный дескриптор Dtot, который рассчитывается с учетом всех атомов в молекуле, оказывается нечувствительным к конформационным изменениям. Величины D для молекул меняются, и направление зависит от типа дескриптора. В случае Dval и Dvdw при переходе от α-спирали к β-структуре наблюдается рост, а для Dunb падение соответствующих величин. Особенно примечательной оказывается молекула аланина, в которой наблюдаются значимые изменения для всех трех фрактальных дескрипторов. Учитывая проведенное ранее нами исследование [14], сдвиги величин D можно связать или с изменением числа атомов, которые используются при расчете, или с изменением точечной группы симметрии молекул при конформационном переходе. Примечательно, что обнаруженный нами ряд из 7 аминокислот (аланин, аспарагин, глицин, гистидин, пролин, серин, триптофан) с заметными изменениями фрактальных дескрипторов практически полностью (за исключением гистидина) попадает в область гидрофобных аминокислот по классификации, использованной в работе [15] ЗАКЛЮЧЕНИЕ Таким образом, анализ полученных результатов свидетельствует о том, что при изменении конформационного состояния аминокислот (α-спираль → β-структура) значимых изменений во фрактальном дескрипторе Dtot, при расчете которого используются все атомы молекулы, не наблюдается. Однако, более специфические дескрипторы Dval, Dvdw и Dunb, которые отражают совокупность валентно связанных, находящихся в Ван-дер-Ваальсовом контакте и несвязанных атомов соответственно, оказываются более чувствительными к конформационному переходу. Для ряда из семи аминокислот (аланин, аспарагин, глицин, гистидин, пролин, серин, триптофан) установлен рост величин Dval, Dvdw и падение величины Dunb. БЛАГОДАРНОСТИ Работа выполнена в рамках государственного задания на 2018 год (тема № 0090-2017-0020). ЛИТЕРАТУРА
|