К 40-летию Института физиологически активных веществ РАН
|
Программа HYBOT 3D MF и её использование в CoMFA
1Институт физиологически активных веществ Российской академии наук
142432 Черноголовка Московской обл., Северный проезд, 1;
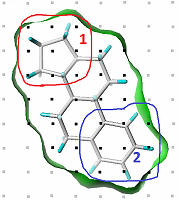
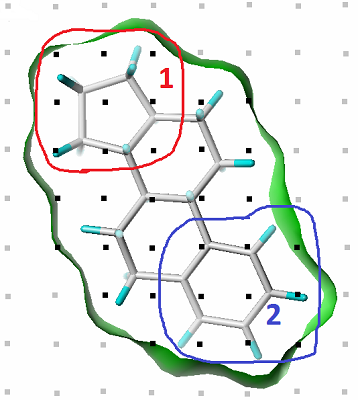
*e-mail: vladlen@ibmh.msk.su Ключевые слова: QSAR; CoMFA; HYBOT; молекулярные поля DOI: 10.18097/BMCRM00073 ВВЕДЕНИЕ Использование различных подходов в области QSAR стало неотъемлемой частью современной вычислительной медицинской химии. Среди них особенно интенсивно развивается группа так называемых 3D методов, когда корреляции с активностями (свойствами) строят на основе определенных характеристик химических соединений, распределённых в пространстве. К таким характеристикам можно отнести различные молекулярные поля, такие как электростатическое и стерическое, используемые в одном из самых первых методов 3D QSAR - методе сравнительного анализа молекулярных полей (Comparative Molecular Field Analysis, CoMFA) [1], имеющем 30-летнюю историю. В данном методе для каждой молекулы согласно общепринятой методике (рис. 1) описывается дискретное молекулярное поле с установленными значениями в узлах трёхмерной решётки, которая описывает либо всё пространство вокруг выровненного набора молекул, либо его отдельную часть. В качестве дескрипторов используются суммы значений для выделенных групп узлов (компонент). Метод CoMFA широко используется и развивается и поныне [2]. На базе аналогичных полей построена также программа GRID (Graphic Retrieval and Information Display) [3,4], в которой есть отличия в определении стерических полей и включены потенциалы водородной связи и гидрофобный потенциал. Потенциалы водородной связи впоследствии появились и в CoMFA, а гидрофобный потенциал можно было добавить как внешнее поле. Метод сравнительного анализа индексов молекулярного подобия (Comparative Molecular Similarity Indices Analysis, CoMSIA) [5,6] представляет собой дальнейшее развитие CoMFA. В данном методе изменена методология расчёта молекулярного поля, когда в узлах решётки рассчитывают индексы молекулярного подобия, и в стандартной версии [SYBYL] имеется возможность использовать электростатические, стерические, гидрофобные поля, а также поля водородных связей. Потенциалы в CoMSIA более плавные. Для поиска корреляций и в CoMFA, и в CoMSIA используется один и тот же стандартный метод статистического анализа PLS (Partial Least Squares или метод частичных наименьших квадратов).
Следует отметить, что наряду с методами описания молекул как набора дискретных дескрипторов, существуют и альтернативные способы, использующие наборы непрерывных гладких функций от пространственных координат и методы машинного обучения с использованием статистических ядер (kernels) [7], но рассматриваемые в работе молекулярные поля были адаптированы и испытаны только для дискретных подходов. В программе GRID для описания водородного связывания используется потенциал «6-8» [8], где изменения энергии при образовании водородной связи описываются следующим набором уравнений:
где Em - оптимальная свободная энергия (ккал/моль), r - расстояние между донором и акцептором водорода (rm – оптимальное, Å).
где t – угол между линиями водород/донор и акцептор/донор (°).
где p – угол между линиями неподелённая пара/акцептор и акцептор/донор (°). При этом устанавливался ограниченный набор пар атомов донор/акцептор для которых были определены оптимальные значения свободной энергии водородной связи и оптимальные расстояния. В работе [9] нами была предложена модификация потенциала «6-8», основанная на факторах водородной связи, рассчитанных программой HYBOT [10]. При этом значения оптимальной свободной энергии водородной связи и оптимального расстояния зависели от произведения акцепторного (Ea) и донорного (Ed) факторов водородного связывания:
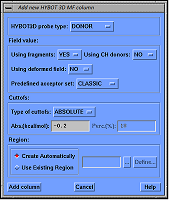
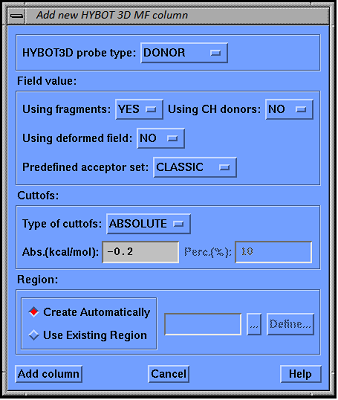
Коэффициенты k4, k7 и k8 были подобраны для каждой пары донор/акцептор сгруппированных по типу химических элементов. Например, k4 = −15, k7 = 0.58, k8 = 2.43 для варианта ‘OH...O’; k4 = −12, k7 = 0.69, k8 = 2.73 для ‘OH...N’; k4 = −10, k7 = 0.94, k8 = 2.80 для ‘NH...N’ и т.д. Используя предложенную модификацию потенциала водородного связывания и молекулу воды в качестве «идеального» зонда можно рассчитать 2 типа молекулярных полей, описывающих акцепторную и донорную способности молекулы соответственно, и использовать эти поля в CoMFA. Соответствующее программное обеспечение, названное «HYBOT 3D MF» было создано и доступно по адресу http://www.ibmc.msk.ru/HYBOT3D. ВОЗМОЖНОСТИ ПРОГРАММЫ «HYBOT 3D MF» Программа «HYBOT 3D MF» написана на языке C++ в виде приложения, исполняемого из командной строки. Тестовая версия не имеет ограничений и доступна для операционной системы Windows 7 и старше. Для интеграции с модулем «QSAR and Advanced CoMFA» пакета SYBYL-Х [11] имеется набор макросов, написанных на SPL (SYBYL Programming Language). Макросы работоспособны и в более старых версиях программы SYBYL. После инсталляции модуля «HYBOT 3D MF» в SYBYL-Х в списке добавления «Computed column» для таблиц появляется опция «HYBOT 3D MF», загружающая окно с параметрами для расчёта молекулярного поля (рис. 2).
Пользователь может выбрать тип поля. Если в качестве пробы используется молекула воды как донор водородного связывания, то поле описывает акцепторную способность молекулы, если же проба трактуется как акцептор водородного связывания – то донорную способность. В качестве параметров поля можно задать варианты расчётов факторов водородного связывания:
Пользователь может установить уровень, ниже которого значение потенциала водородного связывания обнуляется. Данный уровень устанавливается либо в абсолютных значениях в ккал/моль, либо в процентах от максимально возможного. Также пользователь должен определить регион, для которого рассчитывается поле. Автоматически поле рассчитывается для бокса, включающего все молекулы таблицы плюс 4Å по каждой из координат в обоих направлениях. Можно также подгрузить уже имеющийся файл SYBYL, описывающий нужный регион. Помимо описанного способа настройку расчёта поля можно скорректировать через специфический набор переменных TAILOR. Вдобавок к уже описанным опциям имеется возможность определить шаг решётки по умолчанию, а также изменить штрафы, налагаемые за отклонение от идеальных углов (формулы 5 и 6). В стандартном варианте программы GRID эти штрафы пропорциональны квадрату косинуса угла (cos2). В нашей программе допускаются также cos и cos4. После того как колонка с соответствующим полем загружена в таблицу, дальнейший анализ проводится по стандартной для CoMFA процедуре. При загрузке полей имеются некоторые ограничения., а именно: при изменении молекул в ассоциированной с таблицей базе данных автоматический перерасчёт полей HYBOT 3D MF не производится, т.е. процедуру следует запускать по новой. Если ячейка не заполняется, то это может быть связано с тем, что в молекуле имеются незаполненные валентности, либо программа не может привести структуру к стандартному описанию. Последнее можно проверить, записав проблемную молекулу в формате MACCS, отключив возможность записи ароматических связей (тип AR). Если молекула не будет преобразована правильно, то, вероятнее всего, поле HYBOT 3D MF также не будет вычислено. Данный вариант был выбран для того, чтобы избежать ложных результатов. ПРИМЕРЫ ИСПОЛЬЗОВАНИЯ ПРОГРАММЫ «HYBOT 3D MF» Следует отметить, что для того чтобы акцепторные и донорные поля помогли улучшить предсказательную силу QSAR моделей, в наборе молекул не только должны присутствовать потенциальные доноры и/или акцепторы водородного связывания, но и данные группы должны иметь при себе различные химические заместители. Если в группе молекул существует область, в которой у части из них имеется группировка С=O, но при этом в ближайшем окружении данной группы нет других различий (т.е. акцепторная способность не изменяется от молекулы к молекуле), то выигрыша по сравнению с использованием стандартных донорно-акцепторных полей, вероятнее всего, не будет.
В таблице 1 приведен пример использования поля HYBOT 3D MF на выборке из 21 стероида с известной аффинностью к тестостерон связывающему глобулину (стандартный пример SYBYL [1]). Модель 9, использующая оба варианта полей HYBOT 3D MF, демонстрирует наилучшие показатели. Другой пример (табл. 2) – предсказание ингибиторной активности флавоноидов для альдозоредуктазы (EC 1.1.1.21) [12]. Отличительной особенностью данного исследования является то, что были использованы как CoMFA поля, так и CoMSIA. Поскольку эти два метода различаются только на стадии вычисления молекулярного поля, то использование их комбинации в одной модели вполне оправдано. В данном случае явного преимущества полей HYBOT 3D MF при обучении не наблюдается, но имеется выигрыш в предсказательной силе моделей, демонстрируемой на тестовой выборке. Кроме того, при обучении модели без использования полей HYBOT 3D MF дают наилучшие результаты при большем значении параметра «число компонент модели», что может быть причиной переобучения модели.
ЗАКЛЮЧЕНИЕ Подводя итог можно отметить, что для ряда наборов химических соединений, имеющих выраженные различия в части групп с потенциальной возможностью образовывать водородные связи, использование полей HYBOT 3D MF позволяет улучшить предсказательную силу моделей. Несмотря на то, что программа «HYBOT 3D MF» тестировалась, в первую очередь, в составе комплекса SYBYL, её можно применять и в других программах QSAR, использующих для анализа различные молекулярные поля, т.к. по сути это приложение, выполняемое из командной строки, особенности вызова которого описаны в руководстве пользователя. Полноценная версия программы «HYBOT 3D MF» для ОС WINDOWS доступна по адресу http://www.ibmc.msk.ru/HYBOT3D. БЛАГОДАРНОСТИ Работа выполнена в рамках государственного задания на 2018 год (тема № 0090-2017-0020). ЛИТЕРАТУРА
|